Quelle est la différence entre ITS1 et ITS2
Le différence clé Entre ITS1 et ITS2 est que ITS1 est un ADN d'espaceur situé entre 18 et 5.Gènes d'ARNr 8S dans les eucaryotes, tandis que ITS2 est un ADN d'espaceur situé entre 5.Gènes d'ARNr 8s et 28 dans les eucaryotes.
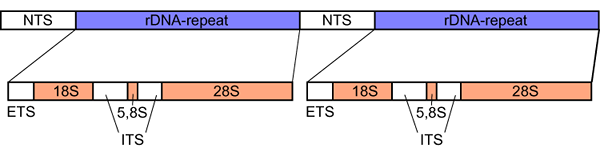
L'espaceur transcrit interne (ITS) est l'ADN de l'espaceur généralement situé entre un petit ARN ribosomal sous-unité et de grands gènes d'ARN ribosomal sous-unité dans le chromosome ou dans la région transcrite correspondante dans la molécule de transcription précurseur d'ARNr polystronic. Dans les bactéries et les archées, il n'y a qu'un seul gènes d'ARNr entre 16S et 23. Mais dans les eucaryotes, il y en a deux ADN d'espaceur: ITS1 (entre 18 et 5.Gènes d'ARNr 8S) et ITS2 (entre 5.8s et 28s ARNr). Par conséquent, ITS1 et ITS2 sont deux ADN d'espaceur trouvés entre les gènes d'ARNr dans les eucaryotes.
CONTENU
1. Aperçu et différence clé
2. Qu'est-ce que son1
3. Qu'est-ce que son2
4. Similitudes - its1 et its2
5. Its1 vs its2 sous forme tabulaire
6. Résumé
Qu'est-ce que son1?
ITS1 est un ADN d'espaceur situé entre 18 et 5.Gènes d'ARNr 8S dans les eucaryotes. Il se réfère à un morceau d'ADN d'espaceur non fonctionnel situé entre les gènes d'ARNr. Son1 a une variation de longueur plus grande que son2. La variation de longueur de ITS1 est approximativement (200-300bp). Ses marqueurs comme ITS1 ont prouvé leur efficacité dans l'élucidation des relations phylogénétiques entre les différents taxons. De plus, l'ADN de l'espaceur est moins conservé que l'ADN d'espaceur ITS2. Ses structures de ses1 ne sont conservées que dans des unités taxonomiques beaucoup plus petites. Chaque grappe ribosomale eucaryote contient également des régions appelées entretoises transcrites externes (5 'et 3' ETS).

Figure 01: Région ITS1
L'ADN d'espaceur ITS1 est très proche de l'espaceur transcrit 5'external (5 'ETS). De plus, si quelqu'un veut amplifier la région ITS1 pour les études de relations phylogénétiques, il doit utiliser une paire d'amorces spécifique (amorces inversées et avant) à cet effet. Pour amplifier la région de sa 1, deux ensembles d'amorces sont normalement utilisés dans les protocoles de PCR des laboratoires. Ils sont son1 en avant et son 2 revers ou son1 Foward et son 4 revers.
Qu'est-ce que son2?
ITS2 est un ADN d'espaceur situé entre 5.Gènes d'ARNr 8s et 28 dans les eucaryotes. Sa région sa2 a une variation de longueur plus courte que la région ITS1. La variation de longueur de ITS2 est environ (180-240bp). Cependant, la région de sa2 est plus conservée que la région de sa 1 dans de nombreuses unités taxonomiques. Il identifie que toutes les séquences de ses2 2 partagent un noyau commun de la structure secondaire. De plus, 40% ITS2 est conservé parmi tous les angiospermes, alors qu'il est possible d'aligner 50% de la région ITS2 à travers la famille et le niveau supérieur.
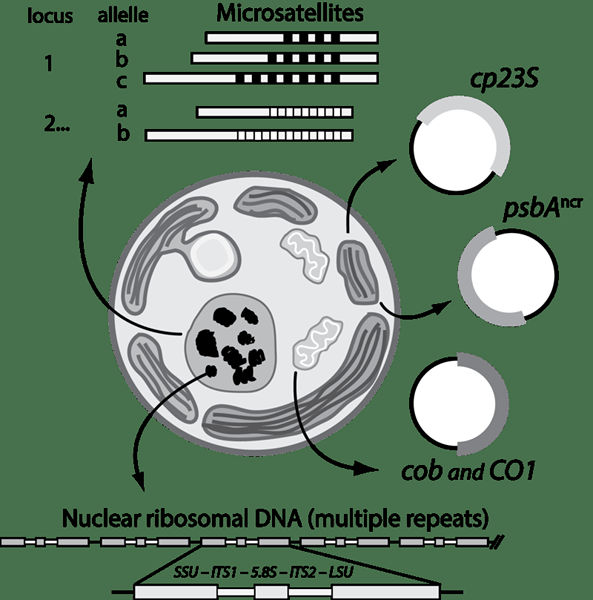
Figure 02: Région ITS2
Quelle que soit la portée de la conservation, l'utilisation de la région de sa2. Pour amplifier la région de sa 2, un ensemble d'amorces spécifique est généralement utilisé dans les protocoles de PCR des laboratoires. C'est son 1 Foward et son 4 revers. Mais lorsque le protocole PCR utilise cet ensemble d'amorces, il amplifie également sa région 1 avec la région ITS2. De plus, l'ADN d'espaceur ITS2 est très proche de l'espaceur transcrit externe (ETS 3 ').
Similitudes entre ITS1 et ITS2
- ITS1 et ITS2 sont deux ADN d'espaceur situés entre les gènes d'ARNr.
- Ils ne sont présents que dans les eucaryotes.
- Les deux peuvent être amplifiés en utilisant des paires d'amorces spécifiques.
- Ils sont tous deux excisés dans la maturation de l'ARNr en tant que sous-produits non fonctionnels.
- Les deux sont très importants pour les études sur les relations phylogénétiques, en particulier dans le codage à barres de l'ADN fongique.
Différence entre ITS1 et ITS2
ITS1 est un ADN d'espaceur présent entre 18s et 5.Gènes d'ARNr 8S dans les eucaryotes tandis que ITS2 est un ADN d'espaceur situé entre 5.Gènes d'ARNr 8s et 28 dans les eucaryotes. C'est donc la principale différence entre ITS1 et ITS2. De plus, ITS1 a une plus grande variation de longueur par rapport à ITS2. D'un autre côté, ITS2 a une variation de longueur plus courte par rapport à ITS1. C'est une autre différence entre ITS1 et ITS2.
L'infographie ci-dessous répertorie les différences entre ITS1 et ITS2 sous forme tabulaire pour une comparaison côte à côte.
Résumé - its1 vs its2
La région interne transcrite (ITS) est un morceau non fonctionnel d'ADN de l'espaceur situé entre les gènes d'ARNr. Dans les bactéries et les archées, il n'y a qu'un seul sa région. Mais dans les eucaryotes, il y en a deux ses régions: its1 et its2. ITS1 est un ADN d'espaceur situé entre 18 et 5.Gènes d'ARNr 8S dans les eucaryotes, tandis que ITS2 est un ADN d'espaceur situé entre 5.Gènes d'ARNr 8s et 28 dans les eucaryotes. Ainsi, c'est le résumé de la différence entre ITS1 et ITS2.
Référence:
1. «Espaceur transcrit interne.«Un aperçu | Sujets ScienceDirect.
2. Schoch, Conrad L., et al. «Nucosomal interne interne interne transcrit la région (ITS) en tant que marqueur de codes à barres ADN universel pour les champignons.»PNAS, Académie nationale des sciences.
Image gracieuseté:
1. «Eucaryot RDNA» par - Pharaon Han supposé - aucune source lisible par machine fournie. Propre travail assumé (sur la base des réclamations du droit d'auteur) (CC BY-SA 3.0) via Commons Wikimedia
2. «MolecularmarkersSymbiodinium» par Allisonmlewis - propre travail (CC BY-SA 4.0) via Commons Wikimedia