Quelle est la différence entre Helix-Loop-Helix et Helix-Turn-Helix
Le différence clé Entre Helix-Loop-Helix et Helix-Turn-Helix Est-ce que l'hélice-boucle-hélice intervient sur la dimérisation des protéines tandis que l'hélice-tour-hélice régule l'expression des gènes par liaison à l'ADN.
Un motif protéique est une séquence courte-conserve associée à des fonctions distinctes de l'ADN. Il est principalement associé à un site structurel spécial à une fonction chimique ou biologique unique. Ces motifs contiennent de petites régions de structures tridimensionnelles d'acides aminés avec différentes molécules de protéines. Habituellement, les motifs individuels ne contiennent que quelques éléments. Helix-Loop-Hélix et Helix-Turn-Hélix contiennent trois éléments. Leurs motifs structurels protéiques comprennent des boucles avec des longueurs variables et des structures non spécifiées.
CONTENU
1. Aperçu et différence clé
2. Qu'est-ce que Helix-Loop-Helix
3. Qu'est-ce que Helix-Turn-Helix
4. Similitudes - Helix-Loop-Hélix et Helix-Turn-Helix
5. Hélice-boucle-hélice vs helix-tour-helix sous forme tabulaire
6. Résumé - Helix-Loop-Helix vs Helix-Turn-Helix
Qu'est-ce que Helix-Loop-Helix?
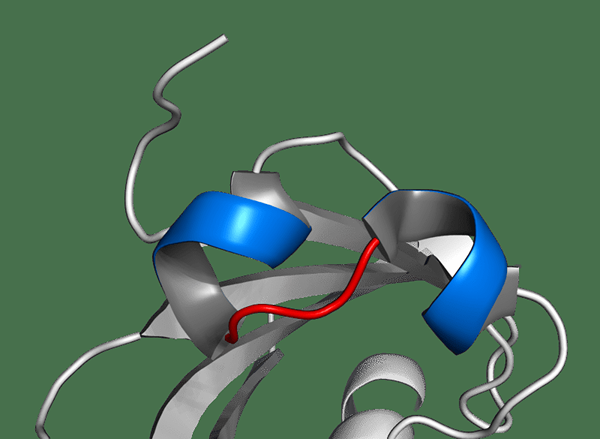
Un hélice-boucle-hélice (HLH) est un motif structurel protéique qui définit l'une des plus grandes familles de facteurs de transcription dimérisants. Ces facteurs de transcription contiennent des résidus d'acides aminés pour faciliter le mécanisme de liaison à l'ADN, et ils sont dimériques. Le motif structurel protéique contient deux hélices α, et elles sont connectées par une boucle. Une hélice apparaît plus petite des deux hélices, et la flexibilité de la boucle permet la dimérisation en emballant et en se repliant contre une autre hélice. L'hélice qui semble plus grande contient généralement des régions de liaison à l'ADN. Les protéines HLH se lient à une séquence consensus connue sous le nom de boîte électronique. Une séquence consensuelle est un ordre calculé contenant des nucléotides ou des résidus d'acides aminés. E-Box est un élément répondant à l'ADN dans certains eucaryotes qui agit comme un site de liaison aux protéines et régule l'expression des gènes.

Figure 01: Motif Helix-Loop-Helix
Les facteurs de transcription HLH sont essentiels pour le développement et l'activité cellulaire. Les protéines HLH appartiennent principalement à six groupes, qui sont indiqués des lettres A à F. Les facteurs de transcription inclus dans chaque groupe sont:
Groupe A: MyOD, MYF5, Beta2 / Neurod1, SCL, P-CAMK, Neurod et Neurogenins,
Groupe B: Max, C-Myc, N-Myc et TCF4
Groupe C: AHR, BMAL-1-Clock, HIF, NPAS1, NPAS3 et MOP5
Groupe D; EMC
Groupe E: Hey1 et Hey2
Groupe F: EBF1
Étant donné que la plupart des facteurs de transcription de HLH sont hétérodimériques, la dimérisation les régule souvent.
Qu'est-ce que Helix-Turn-Helix?
Helix-Turn-Helix (HTH) est un motif structurel protéique capable de lier l'ADN. Chaque monomère est organisé avec deux hélices α et est rejoint par un brin d'acide aminé court. Cela se lie à une rainure dans l'hélice d'ADN. Les motifs HTH régulent généralement l'expression des gènes. La reconnaissance HTH et la liaison à l'ADN sont effectuées par deux hélices α. Une hélice occupe l'extrémité N-terminale tandis que l'autre est à l'extrémité C-terminale. Dans la plupart des scénarios, l'hélice exerce la reconnaissance de l'ADN. Par conséquent, il est connu comme l'hélice de reconnaissance. La liaison à la rainure de l'ADN a lieu à travers une série d'interactions van der Waals et de liaisons hydrogène avec des bases exposées. L'autre hélice α stabilise l'interaction protéique et ADN et ne joue pas un rôle majeur dans la reconnaissance. Cependant, l'hélice de reconnaissance et l'hélice restante ont une orientation similaire.
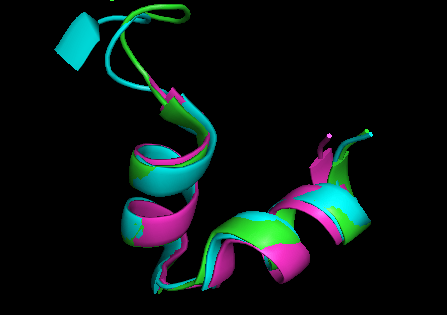
Figure 02: Helix-Turn-Helix de la famille TETR
Le HTH est classé en fonction de la structure et des arrangements spatiaux des hélices. Les principaux types sont dihéliques, tri-hélicoïdaux, tétra-hélicoïdaux et ailés HTH. Le type di-hélicoïdal est le type le plus simple avec deux hélices et un domaine de protéine pliant indépendant. Le type tri-hélicoïdal se trouve dans l'activateur transcriptionnel Myb. Tetra-Hélical Type a une hélice C-terminale supplémentaire. Enfin, le HTH ailé est formé par un faisceau hélicoïdal et une feuille bêta à 3 ou 4 brin.
Quelles sont les similitudes entre Helix-Loop-Helix et Helix-Turn-Helix?
- Hélice-boucle-hélice et hélice-tour-hélice sont des motifs structurels protéiques.
- Les deux contiennent un dénominateur commun dans les facteurs de transcription basaux et spécifiques.
- Ils sont présents dans les eucaryotes.
Quelle est la différence entre Helix-Loop-Helix et Helix-Turn-Helix?
Helix-Loop-Helix Mediate la dimérisation des protéines, tandis que l'hélice-tour-hélice régule l'expression des gènes par liaison à l'ADN. Ainsi, c'est la principale différence entre Helix-Loop-Helix et Helix-Turn-Helix. En outre, HLH contient certains proto-oncogènes et gènes impliqués dans la différenciation codant pour les facteurs de transcription tandis que HTH contient de nombreux gènes homéotiques qui codent pour les facteurs de transcription. De plus, l'hélice-boucle-hélice se compose principalement d'alpha-helices rejoints par une boucle, tandis que Helix-Turn-Helix se compose principalement de boucles rejointes par un court stand d'acides aminés formant un groove.
L'infographie ci-dessous présente les différences entre l'hélice-boucle-hélice et l'hélice-tour-hélice sous forme tabulaire pour une comparaison côte à côte.
Résumé - Helix-Loop-Helix vs Helix-Turn-Helix
Un motif protéique est une séquence courte-conserve associée à des fonctions distinctes de l'ADN. Hélice-boucle-hélice et hélice-tour-hélice sont deux types de motifs structurels protéiques. La principale différence entre l'hélice-boucle-hélice et l'hélice-tour-hélice est que l'hélice-boucle-hélice intervient sur la dimérisation des protéines, tandis que l'hélice-tour-hélice régule l'expression des gènes par liaison à l'ADN. HLH est un motif structurel protéique qui définit l'une des plus grandes familles de facteurs de transcription dimérisants. Le motif structurel protéique contient deux hélices α, et elles sont connectées par une boucle. Hth est un motif structurel protéique capable de lier l'ADN. Chaque monomère est organisé avec deux hélices α, et est rejoint par un brin d'acide aminé court et se lie à une rainure dans l'hélice d'ADN. Donc, cela résume la différence entre Helix-Loop-Helix et Helix-Turn-Helix.
Référence:
1. «Hélix de base en hélice.«Un aperçu | Sujets ScienceDirect.
2. «Helix-Turn-Helix.«Un aperçu | Sujets ScienceDirect.
Image gracieuseté:
1. «Basic Helix Loop Helix» par I, Splette (CC BY-SA 3.0) via Commons Wikimedia
2. “HTH alignment of 3 TetR family members” By Gabbiochem - using PyMol based on PDB: 5DY0 deposited by Palanca & Rubio, PDB: 3VIB deposited by Kumaraswami, Shafer, “Brennan, and PDB: 3ZQL deposited by Le, Schumacher, Lawson, Brennan » Buttner (CC par 3.0) via Commons Wikimedia