Quelle est la différence entre la fin non homologue et la répétition directe homologue
Le Différence clé entre la fin non homologue et la répétition directe homologue Est-ce que la jonction non homologue est une voie qui répare les ruptures à double brin dans l'ADN qui ne nécessite pas de modèle homologue pour guider la réparation, tandis que la répétition directe homologue est une voie qui répare les ruptures à double brin dans l'ADN qui nécessite un modèle homologue pour guider la réparation.
La réparation de l'ADN est un processus où une cellule identifie et corrige les dommages aux molécules d'ADN. Généralement, les activités métaboliques normales et les facteurs environnementaux tels que le rayonnement peuvent causer des dommages à l'ADN. Ces facteurs peuvent entraîner des dizaines de milliers de lésions moléculaires individuelles par cellule par jour. Les voies de réparation des ruptures à double brin d'ADN sont des voies de réparation de l'ADN dans les cellules biologiques. Il y a deux voies de réparation de rupture à double brin d'ADN comme une fin non homologue et une répétition directe homologue.
CONTENU
1. Aperçu et différence clé
2. Quelle est la fin non homologue de rejoindre
3. Qu'est-ce que la répétition directe homologue
4. Similitudes - fin non homologue jointure et répétition directe homologue
5. Fin non homologue rejoignant vs répétition directe homologue sous forme tabulaire
6. Résumé - fin non homologue rejoignant vs répétition directe homologue
Quelle est la fin non homologue de rejoindre?
Fin non homologue se joindre (NHEJ) est une voie qui répare les ruptures de double brin dans l'ADN et ne nécessite pas de modèle homologue pour guider la réparation. Cette voie a été trouvée par Moore et Haber en 1966. Cette voie est généralement guidée par de courtes séquences d'ADN homologues (microhomologies) qui sont souvent présentes dans des surplombs simples aux extrémités des ruptures à double brin. Lorsque les surplombs sont compatibles, la voie NHEJ répare les doubles brins se cassent avec précision. Cependant, lorsque les surplombs ne sont pas parfaitement compatibles, cela entraîne une réparation imprécise qui entraînera une perte de nucléotides. La voie inappropriée NHEJ peut conduire à des translocations, des fusions de télomères et des caractéristiques des cellules tumorales.
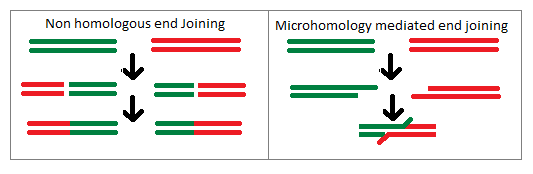
Figure 01: End non homologue rejoignant
NHEJ Pathway a trois étapes principales: la liaison finale et l'attache, le traitement final et la ligature. Chez les mammifères, les protéines appelées MRE11-RAD50-NBS1 (MRN), les ADN-PKC, KU (KU70 et 80) sont impliqués dans le pontage final. L'étape de traitement final implique l'élimination des nucléotides incompatibles ou endommagés et la resynthèse de l'ADN par les ADN polymérases (remplissage de l'écart). L'élimination des nucléotides incompatibles ou endommagées est réalisée par des nucléases telles que Artemis. Les ADN polymérases de la famille X Pol λ et μ chez les mammifères effectuent le remplissage d'espace. Le traitement final n'est pas nécessaire si les extrémités sont déjà compatibles et ont des terminaux 3'hydorxyl ou 5'phosphate. En outre, l'étape de ligature finale est effectuée par le complexe de ligature IV qui se compose d'ADN ligase IV et de son cofacteur XRCC4.
Qu'est-ce que la répétition directe homologue?
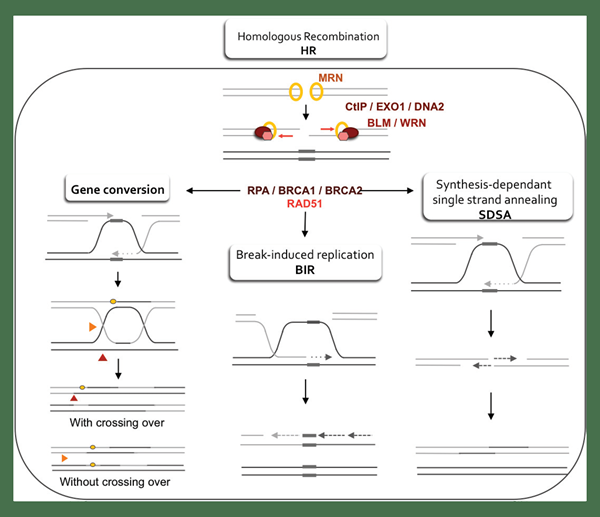
La répétition directe homologue (HDR) est une voie qui répare les ruptures de double brin dans l'ADN avec l'utilisation d'un modèle homologue pour guider la réparation. La manière la plus courante de répétition directe homologue est par la recombinaison homologue. Le mécanisme HDR n'est possible que lorsqu'il y a un morceau homologue d'ADN dans le noyau, principalement dans la phase G2 et S du cycle cellulaire. La voie biologique du HDR commence par la phosphorylation de la protéine histone appelée H2ax dans la zone où se produit la rupture à double brin d'ADN. Cela attire d'autres protéines vers l'emplacement endommagé. Ensuite, le complexe MRN se lie aux extrémités endommagées et empêche les ruptures chromosomiques. Le complexe MRN maintient également les extrémités ensemble. Plus tard, les extrémités de l'ADN sont traitées de telle manière que les résidus inutiles des groupes chimiques sont supprimés et que les surplombs à un brin sont formés.

Figure 02: répétition directe homologue
Chaque morceau d'ADN simple brin est couvert par la protéine appelée RPA, et sa fonction consiste à maintenir des pièces d'ADN à brin simple stable. Après cela, RAD51 remplace la protéine RPA. De plus, lorsque vous travaillez avec BRCA2, RAD51 couple une pièce d'ADN complémentaire qui envahit le brin d'ADN cassé pour former une matrice pour l'ADN polymérase. L'ADN polymérase est maintenue sur l'ADN par une autre protéine connue sous le nom de PCNA. En fin de compte, la polymérase synthétise la partie manquante du brin cassé. De plus, lorsque le brin cassé est resynthétisée, les deux brins doivent à nouveau se découpler. Des modèles de nombreuses façons de découpler sont suggérés. Une fois les brins séparés, le processus est terminé.
Quelles sont les similitudes entre la fin non homologue et la répétition directe homologue?
- Fin non homologue et la répétition directe homologue est deux voies de réparation de rupture à double brin d'ADN.
- Le complexe MRN est impliqué dans les deux voies.
- Les nucléases sont impliquées dans les deux voies.
- Les ADN polymérases sont impliquées dans les deux voies.
- Ces mécanismes peuvent être trouvés dans les procaryotes ainsi que chez les eucaryotes.
- Ce sont tous deux des mécanismes cruciaux pour la survie cellulaire.
Quelle est la différence entre la fin non homologue et la répétition directe homologue?
La jonction de l'extrémité non homologue est une voie qui répare les ruptures à double brin dans l'ADN qui ne nécessite pas de modèle homologue pour guider la réparation, tandis que la répétition directe homologue est une voie qui répare les ruptures à double brin dans l'ADN à l'aide d'un modèle homologue. Ainsi, c'est la principale différence entre la fin non homologue et la répétition directe homologue. En outre, la recombinaison homologue n'est pas impliquée dans la fin de la fin des homologues, tandis que la recombinaison homologue est impliquée dans une répétition directe homologue.
L'infographie ci-dessous présente les différences entre la jonction non homologue et la répétition directe homologue sous forme tabulaire pour une comparaison côte à côte.
Résumé - fin non homologue rejoignant vs répétition directe homologue
La réparation de l'ADN peut être effectuée par différents mécanismes tels que l'inversion directe, la réparation des dommages à un seul brin, la réparation des ruptures à double brin et la synthèse de la translosion. Fin non homologue et les répétitions directes homologues sont deux voies de réparation de rupture à double brin d'ADN. La jointure de fin non homologue ne nécessite pas de modèle homologue pour guider la voie de réparation de l'ADN. La répétition directe homologue est une voie qui nécessite un modèle homologue pour guider la réparation de l'ADN. C'est donc la principale différence entre la fin non homologue et la répétition directe homologue.
Référence:
1. «Fin non homologue rejoignant.«Un aperçu | Sujets ScienceDirect.
2. «Réparation dirigée par l'homologie.«Un aperçu | Sujets ScienceDirect.
Image gracieuseté:
1. «Fin non homologue de jointure et de microhomologie Médiate de fin de jointure» par Cynth3004 à English Wikipedia (CC BY-SA 3.0) via Commons Wikimedia
2. «Modèles de réparation de rupture à double brin qui agissent via une recombinaison homologue» de Camille Gelot, Indiana Magdalou et Bernard S. Lopez - https: // doi.org / 10.3390 / Genes6020267 (CC par 4.0) via Commons Wikimedia