Différence entre QTL et GWAS
Le différence clé entre QTL et GWAS s'appuie sur le type de séquences utilisées dans l'analyse. QTL utilise des loci de gènes de liaison pour analyser les traits phénotypiques associés à l'héritage polygénique tandis que GWAS utilise des séquences du génome entier pour analyser.
Les cartes QTL et les GWAS jouent un rôle important dans les analyses génétiques pour différents traits. De plus, ils sont importants pour analyser diverses conditions de maladie avec une étiologie inconnue. De plus, le séquençage joue un rôle clé dans les deux techniques. Ces techniques peuvent en outre être associées à d'autres techniques de débit élevé pour une précision et une précision accrues.
CONTENU
1. Aperçu et différence clé
2. Qu'est-ce que QTL
3. Qu'est-ce que GWAS
4. Similitudes entre QTL et GWAS
5. Comparaison côte à côte - QTL vs gwas sous forme tabulaire
6. Résumé
Qu'est-ce que QTL?
QTL représente Locus de traits quantitatifs. C'est une région de l'ADN associée à un trait phénotypique. Généralement, un QTL donne lieu à des effets polygéniques. La distribution des QTL varie et le nombre de QTLS suggère le degré de variation d'un trait phénotypique particulier. De plus, ils codent généralement des traits continus sous-tendez et non des traits discrets.
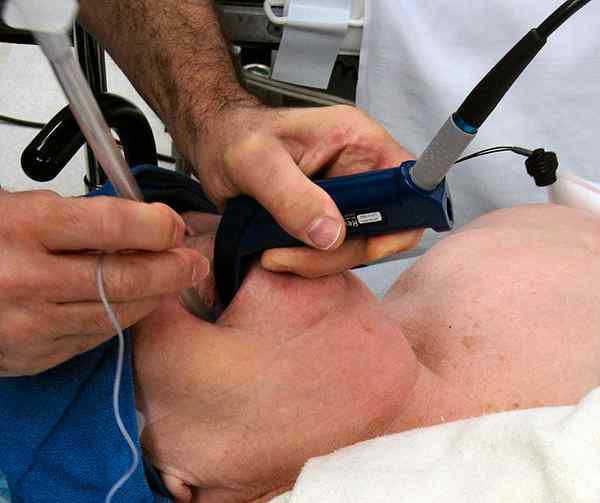
Lors de l'identification des régions QTL, il est important de séquencer une zone QTL particulière. De plus, pour faciliter les recherches et la recherche, le séquençage et le stockage des données de séquence des régions QTL communes ont lieu avec l'implication de la bioinformatique. Nous appelons cette technique QTL Mapping. Par la suite, une base de données s'accumule avec les séquences de la région QTL.

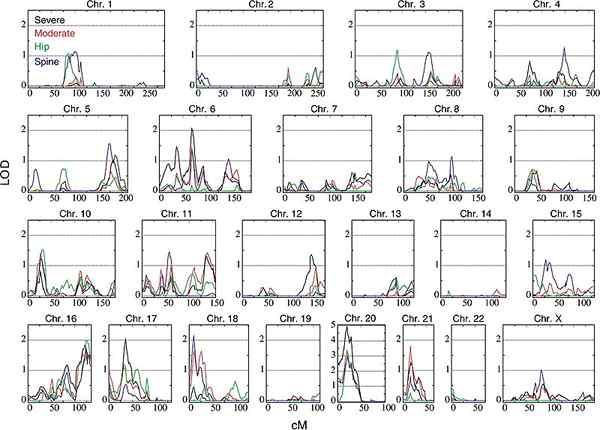
Figure 01: scan QTL
De plus, les applications des séquences QTL reposent principalement sur le Blast Tool, où la similitude des séquences peut être déterminée. Il est important de déduire les relations entre les organismes. De plus, il est important de déterminer la complexité d'un phénotype polygénique particulier. Il détermine également l'évolution d'un trait particulier.
À l'heure actuelle, l'analyse QTL est combinée avec d'autres techniques de débit élevé comme les puces à ADN. Ceci est important dans l'analyse d'expression du phénotype. Actuellement, les scientifiques sont très intéressés à développer la base de données QTL, car les régions QTL sont responsables de nombreux traits importants de différentes espèces.
Qu'est-ce que GWAS?
Gwas représente Étude de l'association large du génome. Il se réfère également aux études de l'association du génome entier. Ces études se concentrent principalement sur les études d'observation. Il analyse les variantes génétiques de différentes personnes généralement associées à un trait spécifique. De plus, l'ensemble du génome est important pour l'analyse GWAS.
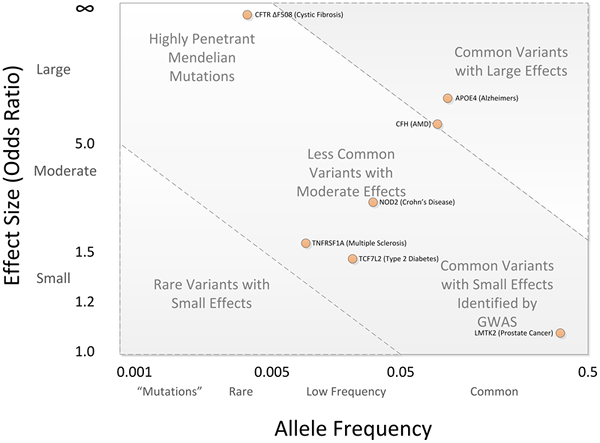
Figure 02: GWAS
GWAS est un outil important dans l'analyse des polymorphismes mononucléotidiques associés à diverses affections pathologiques. Il s'agit également d'une étude comparative des différents polymorphismes un seul nucléotide à travers une large population. De plus, l'échantillon d'étude de GWAS est très élevé; Par conséquent, il prend également le format d'une étude de cohorte transversale.
De plus, la première étude GWAS a eu lieu en ce qui concerne l'infarctus du myocarde et l'analyse des gènes associés à l'infarctus du myocarde. À l'heure actuelle, GWAS joue un rôle important dans la détermination du contexte génétique des maladies complexes avec une étiologie inconnue.
Quelles sont les similitudes entre QTL et GWAS?
- Ils s'appuient sur des données sur les séquences d'ADN pour l'analyse.
- Les deux sont associés aux arrière-plans génétiques de divers caractères.
- De plus, ils nécessitent des outils de bioinformatique pour déduire et interpréter les résultats.
- Ils se font sur de grandes populations.
Quelle est la différence entre QTL et GWAS?
QTL analyse les traits phénotypiques associés. Ainsi, c'est la principale différence entre QTL et GWAS. La cartographie QTL est relativement une technique moins complexe que GWAS car elle nécessite un séquençage du génome entier.
L'infographie ci-dessous résume la différence entre QTL et GWAS sous forme tabulaire.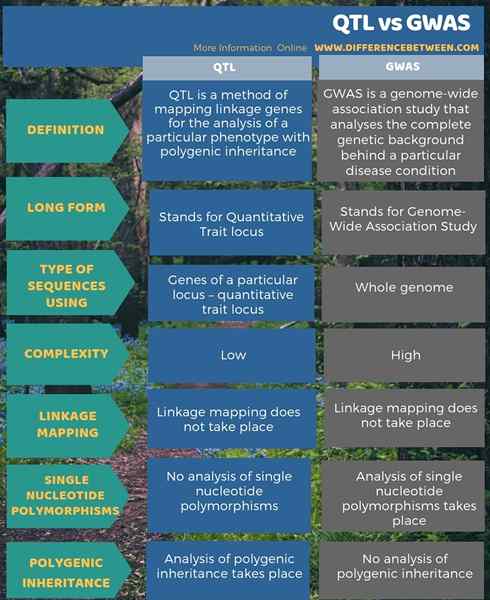
Résumé - QTL vs GWAS
La cartographie QTL et les GWAS jouent un rôle majeur dans la détermination du contexte génétique de divers traits phénotypiques. Ils aident également à analyser le contexte génétique de différentes maladies. La cartographie QTL mappe les gènes de liaison responsables des traits continus et implique également la cartographie des gènes de l'héritage polygénique. GWAS, en revanche, se déroule avec une analyse du génome entier pour les polymorphismes un seul nucléotide. De plus, cela se déroule dans une population plus importante. Ainsi, c'est le résumé de la différence entre QTL et GWAS.
Référence:
1. Liu, Ruixien, et al. «L'analyse GWAS et l'identification QTL des traits de qualité des fibres et des composants de rendement dans le coton des hautes terres en utilisant des marqueurs SNP à haute densité enrichis.”Frontiers in Plant Science, Frontiers Media S.UN., 13 septembre. 2018, disponible ici.
2. «Gwas vs. Mappage QTL?”Derniers articles, disponibles ici.
Image gracieuseté:
1. «Exemple d'un QTL-scan à l'échelle du génome de la biologie PLOS» par - Styrkarsdottir U, Cazier JB, Kong A, Rolfsson O, Larsen H, et al. (2003) Lien de l'ostéoporose au chromosome 20p12 et association à BMP2. PLOS BIOL 1 (3): E69 (CC par 2.5) Via Commons Wikimedia
2. «Effets des allèles de la maladie de GWAS» par Bush WS et Moore JH - Bush WS, Moore JH (2012) Chapitre 11: Études d'association à l'échelle du génome. PLOS Comput Biol 8 (12): E1002822. doi: 10.1371 / Journal.PCBI.1002822, (CC par 2.5) Via Commons Wikimedia