Quelle est la différence entre CGH et Array CGH
Le différence clé Entre CGH et Array CGH est que CGH est une méthode cytogénétique moléculaire traditionnelle pour analyser la variation du nombre de copies dans l'ADN d'un échantillon de test qui a une résolution plus faible, tandis que le tableau CGH est une méthode cytogénétique moléculaire entièrement automatisée pour analyser la variation du nombre de copies dans l'ADN d'un échantillon de test qui a un plus haute résolution.
CGH et Array CGH sont deux techniques pour détecter les variations du nombre de copies chez les individus. La variation du nombre de copies fait référence aux sections du génome qui sont répétées. Le nombre de copies varie entre les individus. La variation du nombre de copies est généralement classée en deux groupes: les répétitions courtes et les longues répétitions. L'un des exemples les plus connus pour une courte variation du nombre de copies est la répétition du trinucléotide CAG dans le gène Huntingtin. Cela provoque un trouble neurologique appelé Huntingtin. Le meilleur exemple pour les longues répétitions la variation du nombre de copies est le gène alpha amylase, qui provoque une résistance à l'insuline. La variation du nombre de copies peut être détectée par des techniques telles que Fish, CGH et Array CGH.
CONTENU
1. Aperçu et différence clé
2. Qu'est-ce que CGH
3. Qu'est-ce que Array CGH
4. Similitudes - CGH et Array CGH
5. CGH vs Array CGH sous forme tabulaire
6. Résumé - CGH vs Array CGH
Qu'est-ce que CGH?
CGH est une méthode cytogénétique moléculaire traditionnelle pour analyser la variation du nombre de copies dans l'ADN d'un échantillon de test par rapport à un échantillon de référence. Il a une résolution plus faible par rapport aux techniques automatisées. L'objectif de cette technique est de comparer rapidement et efficacement deux échantillons d'ADN génomique qui découlent de différentes sources.
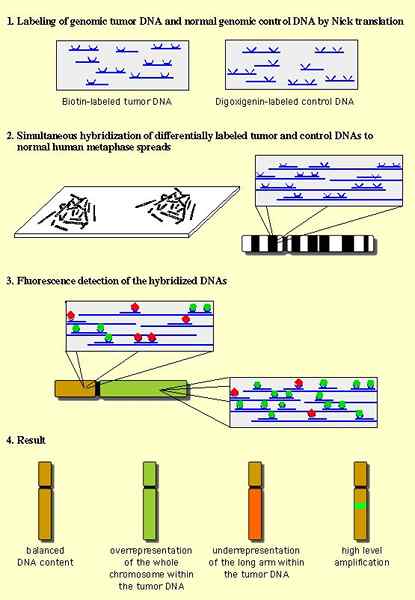
Figure 01: CGH
La procédure implique l'isolement de l'ADN de deux sources: test et référence. Ensuite, l'étape suivante est le marquage indépendant des échantillons d'ADN de test et de référence avec des fluorophores de différentes couleurs. Plus tard, les deux échantillons d'ADN ont été dénaturés afin d'obtenir les échantillons d'ADN simple brin. Enfin, l'hybridation des deux échantillons d'ADN résultants dans un rapport 1: 1 à une propagation de métaphase normale des chromosomes a été réalisée. Les différences chromosomiques peuvent être identifiées entre les deux sources à l'aide d'un microscope à fluorescence et d'un logiciel informatique. De plus, la technique CGH est uniquement capable de détecter les anomalies chromosomiques déséquilibrées.
Qu'est-ce que Array CGH?
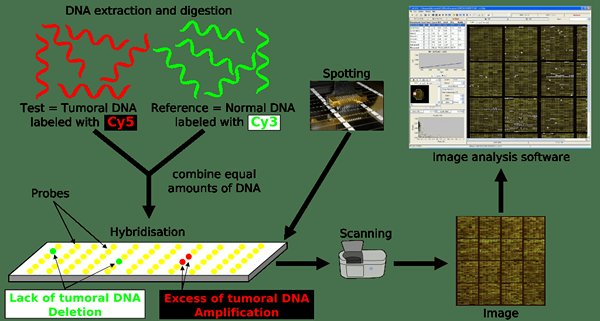
Array CGH est une méthode cytogénétique moléculaire entièrement automatisée pour analyser la variation du nombre de copies dans l'ADN d'un échantillon de test par rapport à un échantillon de référence. Il a une résolution plus élevée, et il a été développé afin de minimiser les limites du CGH traditionnel. Array CGH combine les principes de CGH avec l'utilisation de puces à ADN. Au lieu d'utiliser la propagation des chromosomes de métaphase, cette méthode utilise des lames réparties avec de petits segments d'ADN comme cibles. Ces microréseaux ont été fabriqués en déposant et en immobilisant une petite quantité d'ADN (sondes) sur un support en verre solide. La taille des sondes peut varier de l'oligonucléotide à un chromosome artificiel bactérien.

Figure 02: Array CGH
Quelles que soient les sondes, la méthode de base du tableau CGH est similaire à CGH. Une fois que les sondes contenant des microréseaux sont préparées, les deux échantillons d'ADN (test et référence) marqués avec des fluorophores de différentes couleurs sont mélangés et ajoutés au pudonnage. Étant donné que l'ADN dans les échantillons est simple brin en raison de la dénaturation, cet ADN s'hybride avec des sondes simples à brin. À l'aide d'un système d'imagerie numérique, il est possible d'identifier les différences chromosomiques entre les deux sources.
Quelles sont les similitudes entre CGH et Array CGH?
- CGH et Array CGH sont deux techniques pour détecter les variations de numéro de copie.
- Ce sont des méthodes cytogénétiques moléculaires.
- Les deux méthodes diagnostiquent les anomalies chromosomiques déséquilibrées.
- Les fluorophores sont impliqués dans les deux méthodes.
- Les deux méthodes peuvent être utilisées pour détecter les troubles génétiques en raison des variations du nombre de copies.
- Ils sont largement utilisés dans l'analyse génomique comparative.
Quelle est la différence entre CGH et Array CGH?
Le CGH est une méthode cytogénétique moléculaire traditionnelle pour analyser la variation du nombre de copies dans l'ADN d'un échantillon de test qui a une résolution plus faible, tandis que le tableau CGH est une méthode cytogénétique moléculaire entièrement automatisée pour analyser la variation du nombre de copies dans l'ADN d'un échantillon de test qui a une résolution plus élevée. Donc, c'est la principale différence entre CGH et Array CGH. De plus, dans CGH, les échantillons d'ADN de test et de référence s'hybrident avec une propagation normale de métaphase des chromosomes, mais dans le tableau CGH, les échantillons d'ADN de test et de référence s'hybrident avec les sondes d'ADN immobilisées contenant des microréseaux.
L'infographie ci-dessous répertorie les différences entre CGH et le tableau CGH sous forme tabulaire pour une comparaison côte à côte.
Résumé - CGH vs Array CGH
CGH et Array CGH sont deux techniques cytogénétiques moléculaires pour détecter les anomalies chromosomiques déséquilibrées. Ils sont largement utilisés dans l'analyse génomique comparative. CGH est une méthode cytogénétique moléculaire traditionnelle pour analyser la variation du nombre de copies dans l'ADN d'un échantillon de test qui a une résolution plus faible. En revanche, Array CGH est une méthode cytogénétique moléculaire entièrement automatisée pour analyser la variation du nombre de copies dans l'ADN d'un échantillon de test qui a une résolution plus élevée. Ainsi, cela résume la différence entre CGH et Array CGH.
Référence:
1. «Hybridation génomique comparative.«Un aperçu | Sujets ScienceDirect.
2. Heather, James M., et la chaîne Benjamin. «La séquence de séquenceurs: l'histoire de l'ADN de séquençage.»Genomics, Academic Press, 10 novembre. 2015.
Image gracieuseté:
1. «CGH Schema» par Mbaudis (Talk) (Uploads) - Propre travaux (CC par 2.5) Via Commons Wikimedia
2. «Protocole Array-CGH» par Philippe Hupé - Emmanuel Barillot, Laurence Calzone, Philippe Hupé, Jean-Philippe Vert, Andrei Zinovyev, Computational Systems Biology of Cancer Chapman & «Hall / CRC Mathématique» Biology Computational Biology, 2012 (CC BY-SA 3.0) via Commons Wikimedia