Différence entre les puces à ADN et le séquençage d'ARN
Différence clé - ADN ADN vs ARN séquençage
Le transcriptome représente l'ensemble du contenu de l'ARN présent dans une cellule comprenant l'ARNm, l'ARNr, l'ARNt, l'ARN dégradé et l'ARN non dégradé. Le transcriptome de profilage est un processus important afin de comprendre les informations cellulaires. Il existe plusieurs méthodes avancées pour le profilage du transcriptome. Les microréseaux et le séquençage d'ARN sont deux types de technologies développées pour analyser le transcriptome. La principale différence entre les microréseaux et le séquençage d'ARN est que Le microréseau est basé sur le potentiel d'hybridation des sondes marquées prédésignées avec des séquences d'ADNc cibles tandis que le séquençage d'ARN est basé sur le séquençage direct des brins d'ADNc par des techniques de séquençage avancées telles que NGS. Un microréseau est effectué avec les connaissances antérieures sur les séquences et le séquençage d'ARN est effectué sans les connaissances antérieures sur les séquences.
CONTENU
1. Aperçu et différence clé
2. Qu'est-ce que les microréseaux
3. Qu'est-ce que le séquençage d'ARN
4. Comparaison côte à côte - Aicroréseau vs séquençage d'ARN
5. Résumé
Qu'est-ce que les microréseaux?
ADN est une méthode de débit robuste, fiable et à haut débit utilisée pour le profilage du transcriptome par les scientifiques. C'est l'approche la plus populaire pour l'analyse des transcrits. Il s'agit d'une méthode à faible coût, qui dépend des sondes d'hybridation.
La technique commence par l'extraction de l'ARNm de l'échantillon et la construction de la bibliothèque d'ADNc à partir de l'ARN total. Ensuite, il est mélangé à des sondes prédésignées marquées par fluorescence sur une surface solide (matrice de spot). Les séquences complémentaires hybrident avec les sondes marquées dans le microréseau. Ensuite, les microréseaux sont lavés et dépistés, et l'image est quantifiée. Les données recueillies doivent être analysées pour obtenir les profils d'expression relatifs.
L'intensité des sondes de microréseaux est supposée proportionnelle à la quantité de transcriptions dans l'échantillon. Cependant, la précision de la technique dépend des sondes conçues, de la connaissance préalable de la séquence et de l'affinité des sondes pour l'hybridation. Par conséquent, la technologie des microréseaux a des limites. La technique de microréseaux ne peut pas être effectuée avec des transcriptions de faible abondance. Il ne fait pas différencier les isoformes et identifier les variantes génétiques. Étant donné que cette méthode dépend de l'hybridation des sondes, certains problèmes liés à l'hybridation tels que l'hybridation croisée, l'hybridation non spécifique, etc. se produit dans la technique des puces à ADN.

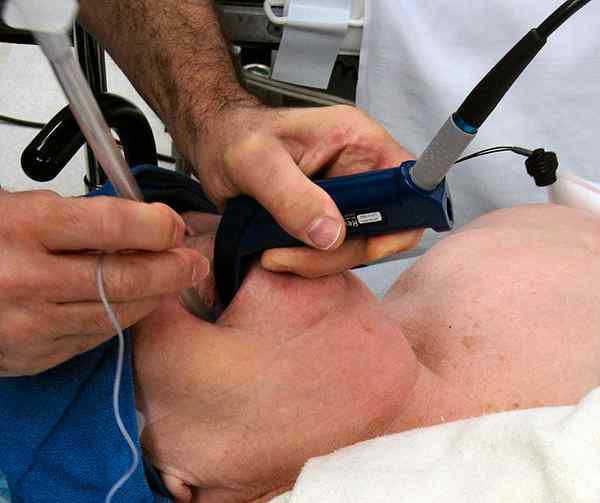
Figure 01: ADN
Qu'est-ce que le séquençage d'ARN?
Séquençage de fusil de chasse d'ARN (ARN SEQ) est une technique de séquençage du transcriptome entier récemment développé. C'est une méthode de débit rapide et à haut débit de profilage du transcriptome. Il quantifie directement l'expression des gènes et se traduit par une étude approfondie du transcriptome. L'ARN SEQ ne dépend pas des sondes prédéfinies ou de la connaissance préalable des séquences. Par conséquent, la méthode ARN SEQ a une sensibilité et une capacité élevées à détecter de nouveaux gènes et variantes génétiques.
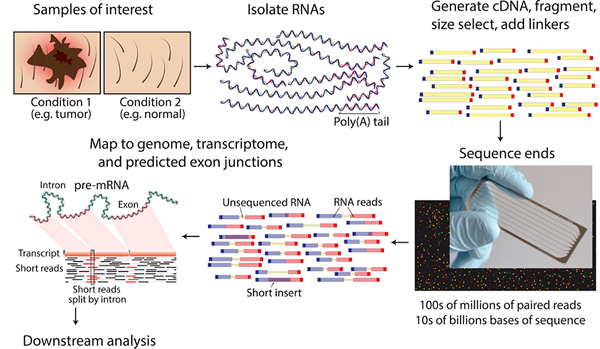
La méthode de séquençage d'ARN est effectuée via plusieurs étapes. L'ARN total de la cellule doit être isolé et fragmenté. Ensuite, en utilisant la transcriptase inverse, une bibliothèque d'ADNc doit être préparée. Chaque brin d'ADNc doit être ligaturé avec des adaptateurs. Ensuite, les fragments ligaturés doivent être amplifiés et purifiés. Enfin, en utilisant une méthode NGS, le séquençage de l'ADNc doit être effectué.

Figure 02: séquençage d'ARN
Quelle est la différence entre les puces à ADN et le séquençage d'ARN?
Séquençage de microréseau vs ARN | |
| ADN est une méthode de débit robuste, fiable et à haut débit. | Le séquençage de l'ARN est une méthode précise et à haut débit. |
| Coût | |
| Ceci est une méthode à faible coût. | Ceci est une méthode coûteuse. |
| Analyse d'un grand nombre d'échantillons | |
| Cela facilite l'analyse simultanément un grand nombre d'échantillons. | Cela facilite l'analyse d'un grand nombre d'échantillons. |
| L'analyse des données | |
| L'analyse des données est complexe. | Plus de données sont générées dans cette méthode; Par conséquent, le processus est plus complexe. |
| Connaissance antérieure des séquences | |
| Cette méthode est basée sur des sondes d'hybridation, donc une connaissance préalable des séquences dans requise. | Cette méthode ne dépend pas des connaissances de séquence antérieure. |
| Variations structurelles et nouveaux gènes | |
| Cette méthode ne peut pas détecter les variations structurelles et les nouveaux gènes. | Cette méthode peut détecter des variations structurelles telles que la fusion des gènes, l'épissage alternatif et les nouveaux gènes. |
| Sensibilité | |
| Cela ne peut pas détecter les différences d'expression des isoformes, donc cela a une sensibilité limitée. | Cela a une sensibilité élevée. |
| Résultat | |
| Cela ne peut entraîner que des niveaux d'expression relatifs. Cela ne donne pas de quantification absolue de l'expression des gènes. | Il donne des niveaux d'expression absolues et relatives. |
| Réanalyse des données | |
| Cela doit être redimensionné pour réanalyser. | Les données de séquençage peuvent être réanalysées. |
| Besoin de personnel et d'infrastructure spécifiques | |
| Les infrastructures et le personnel spécifiques ne sont pas requis pour les puces à ADN. | Infrastructure spécifique et personnel requis par le séquençage d'ARN. |
| Problèmes techniques | |
| La technique de puces à ADN a des problèmes techniques tels que l'hybridation croisée, l'hybridation non spécifique, le taux de détection limité des sondes individuelles, etc. | La technique d'ARN SEQ évite les problèmes techniques tels que l'hybridation croisée, l'hybridation non spécifique, le taux de détection limité des sondes individuelles, etc. |
| Les préjugés | |
| Il s'agit d'une méthode biaisée car elle dépend de l'hybridation. | Le biais est faible par rapport aux puces à ADN. |
Résumé - ADN ADN contre ARN le séquençage
Les méthodes de séquençage de puces à ARN et d'ARN sont des plateformes de débit élevés développées pour le profilage du transcriptome. Les deux méthodes produisent des résultats fortement corrélés aux profils d'expression génique. Cependant, le séquençage de l'ARN a des avantages par rapport aux microréseaux pour l'analyse de l'expression des gènes. Le séquençage d'ARN est une méthode plus sensible pour la détection de transcrits à faible abondance que les puces à ADN. Le séquençage d'ARN permet également la différenciation entre les isoformes et l'identification des variantes de gènes. Cependant, les microréseaux sont le choix courant de la plupart des chercheurs, car le séquençage de l'ARN est une technique nouvelle et coûteuse avec des défis de stockage de données et une analyse des données complexes.
Les références:
1.Wang, Zhong, Mark Gerstein et Michael Snyder. «RNA-seq: un outil révolutionnaire pour la transcriptomique.»Nature Reviews. La génétique. U.S. Bibliothèque nationale de médecine, Jan. 2009. la toile. 14 mars. 2017
2.Rogler, Charles E., Tatyana Tchaikovskaya, Raquel Norel, Aldo Massimi, Christopher Plescia, Eugeny Rubashevsky, Paul Siebert et Leslie E. Rogler. «Les microréseaux d'expression de l'ARN (REM), une méthode à haut débit pour mesurer les différences d'expression des gènes dans divers échantillons biologiques.»Recherche des acides nucléiques. Oxford University Press, 01 janvier. 2004. la toile. 15 mars. 2017
3.Zhao, Shanrong, Wai-Ping Fung-Leung, Anton Bittner, Karen Ngo et Xuejun Liu. «Comparaison de l'ARN-seq et des microréseaux dans le profilage du transcriptome des cellules T activées.»Plos un. Bibliothèque publique des sciences, Jan. 2014. la toile. 15 mars. 2017
Image gracieuseté:
1. "Journal.PCBI.1004393.G002 ”par Malachi Griffith, Jason R. Walker, Nicholas C. Espions, Benjamin J. Ainscough, obi l. Griffith - (CC par 2.5) Via Commons Wikimedia
2. «Microarray» de Bill Branson (photographe) - National Cancer Institute (Domain public) via Commons Wikimedia