Différence entre Maxam Gilbert et Sanger Sequencing
Différence clé - MAXAM GILBERT VS SANGER SEQUENCING
Les nucléotides sont les unités structurelles de base et les éléments constitutifs de l'ADN. La molécule d'ADN est composée d'une chaîne polynucléotidique. Il y a quatre nucléotides différents trouvés dans l'ADN. Ces nucléotides sont composés de quatre bases azotées différentes nommées (adénine), g (guanine), c (cytosine), t (thymine). L'ordre des nucléotides dans la molécule d'ADN a une grande importance car il code pour une information génétique importante pour la croissance et le développement des organismes. Le séquençage d'ADN fait référence au processus qui détermine la séquence nucléotidique précise de l'ADN. Il existe différentes méthodes de séquençage d'ADN. Le séquençage de Maxam Gilbert et le séquençage d'ADN Sanger sont deux méthodes de séquençage d'ADN qui appartiennent au séquençage de première génération. La procédure de séquençage de Maxam Gilbert détermine la séquence de base en clivant chimiquement les fragments d'ADN marqués à 5 '. La procédure de séquençage Sanger détermine la séquence nucléotidique en synthétisant l'ADN simple brin en utilisant l'ADN polymérase et les didésoxynucléotides et l'électrophorèse sur gel. C'est la principale différence entre Maxam Gilbert et le séquençage de Sanger.
CONTENU
1. Aperçu et différence clé
2. Qu'est-ce que Maxam Gilbert
3. Qu'est-ce que le séquençage Sanger
4. Comparaison côte à côte - MAXAM GILBERT VS SANGER SEQUENCING
5. Résumé
Qu'est-ce que le séquençage Maxam Gilbert?
Séquençage de Maxam Gilbert, également connu sous le nom Méthode de séquençage chimique, est une technique qui a été développée pour déterminer l'ordre des nucléotides dans l'ADN. Cette méthode a été introduite par Walter Gilbert et Alan Maxam en 1976 et est devenue populaire car elle peut être réalisée directement avec de l'ADN purifié. La méthode Maxam Gilbert appartient à la première génération de séquençage d'ADN, et c'était la première méthode de séquençage largement utilisée par les scientifiques.
Le principe de base de cette méthode réside dans la restriction des fragments d'ADN marqués à l'extrémité à des bases spécifiques par des produits chimiques et des conditions spécifiques à la base et la séparation des fragments marqués par électrophorèse comme le montre la figure 01. Les fragments sont séparés en fonction de leurs tailles sur le gel. Étant donné que les fragments sont étiquetés, la séquence de la molécule d'ADN peut être facilement déduite.
La méthode Maxam Gilbert utilise des produits chimiques spécifiques de base pour briser l'ADN à des bases spécifiques. Deux produits chimiques communs nommés diméthyl sulfate et les produits chimiques de l'hydrazine sont utilisés pour attaquer sélectivement les purines et les pyrimidines, respectivement.
La méthode de séquençage Maxam Gilbert est effectuée via plusieurs étapes comme suit.
- Purification de la séquence d'ADN à l'aide d'endonucléases de restriction
- Étiquetage des extrémités des fragments d'ADN en ajoutant des phosphates radioactifs
- Purification des fragments marqués à partir de fragments non marqués par électrophorèse sur gel
- Séparation de l'ADN marqué en fin
- Électrophorèse du contenu de chaque tube sur des lignes séparées sur un gel et une séparation des fragments selon leur longueur.
- Détection des fragments par autoradiographie.
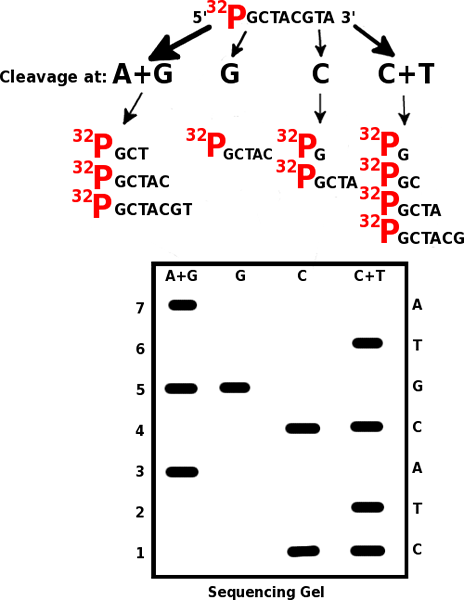
Figure 01: Séquençage Maxam Gilbert
Qu'est-ce que le séquençage Sanger?
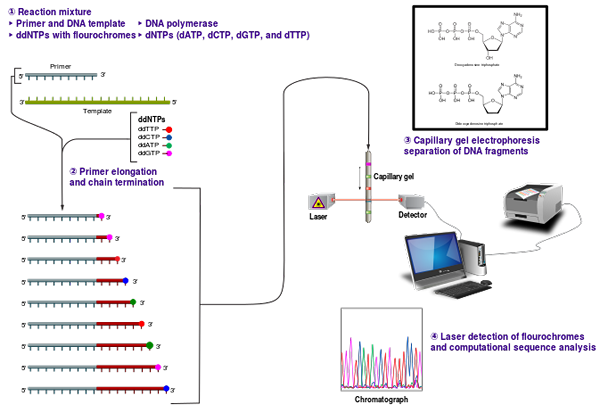
Le séquençage de Sanger est une méthode de séquençage développée par Frederick Sanger et ses collègues en 1977 pour déterminer la séquence de base d'un fragment d'ADN donné. Il est également connu sous le nom séquençage de terminaison de la chaîne ou Méthode de séquençage de la didésoxy. Cette méthode fonctionne sur le principe de l'incorporation sélective des didésoxynucléotides (DDNTP), tels que DDGTP, DDCTP, DDATP et DDTTP). Les didésoxynucléotides manquent de 3 'groupes OH pour la formation de liaisons phosphodiester avec un nucléotide adjacent. Par conséquent, la formation de brin s'arrête une fois qu'un DDNTP est incorporé dans le volet nouvellement formant pendant le séquençage Sanger.
Dans cette méthode, quatre réactions de synthèse d'ADN distinctes (PCR) sont effectuées dans quatre tubes séparés avec un type de DDNTP. D'autres exigences sont également fournies pour les tubes pour la PCR, y compris les amorces, les DNTP, la polymérase Taq, les conditions spécifiques, etc. Quatre réactions distinctes sont effectuées en quatre tubes avec quatre mélanges. Après les réactions de PCR, les fragments d'ADN résultants sont dénaturés par la chaleur et séparés par électrophorèse sur gel. Ensuite, les fragments sont visualisés en utilisant une amorce étiquetée (radioactive ou fluorescent) ou DNTP comme indiqué sur la figure 02.

Figure 02: séquençage Sanger
Quelle est la différence entre Maxam Gilbert et le séquençage de Sanger?
MAXAM GILBERT VS Sanger Séquençage | |
| Le séquençage de Maxam Gilbert est la première technique développée pour le séquençage de l'ADN. | La méthode de séquençage de Sanger a été introduite après la méthode de séquençage de Maxam Gilbert. |
| Usage | |
| Cette méthode est rarement utilisée. | Le séquençage Sanger est systématiquement utilisé pour le séquençage. |
| Utilisation de produits chimiques dangereux | |
| Il utilise des produits chimiques dangereux. | L'utilisation de produits chimiques dangereux est limité par rapport à la méthode Maxam Gilbert. |
| Étiquetage pour la détection | |
| Cette méthode utilise le p radioactif32 pour étiqueter les extrémités des fragments d'ADN. | Le séquençage de Sanger utilise des DDNTP marqués par radio et fluorescence. |
Résumé - MAXAM GILBERT VS Sanger Séquençage
Le séquençage de Maxam Gilbert et Sanger est deux types de techniques de séquençage d'ADN qui sont sous séquençage d'ADN de première génération. Le séquençage de Maxam Gilbert est la première méthode introduite pour le séquençage de l'ADN en 1976, et elle est réalisée en brisant les fragments d'ADN marqués par fin par des produits chimiques spécifiques à la base. Par conséquent, il est connu sous le nom de séquençage chimique. La méthode de séquençage Sanger a été introduite en 1977 et est basée sur les réactions de terminaison de la chaîne entraînée par DDNTP. Méthode de séquençage Sanger populaire que la méthode Maxam Gilbert en raison de plusieurs inconvénients de la méthode Maxam Gilbert tels que la consommation excessive de temps, l'utilisation de produits chimiques dangereux, etc. C'est la différence entre Maxam Gilbert et le séquençage de Sanger.
Les références:
1.Maxam, un. M et Walter Gilbert. «Une nouvelle méthode pour séquencer l'ADN."Actes de l'Académie nationale des sciences. National Acad Sciences, 9 décembre. 1976. la toile. 30 mars. 2017
2.Heather, James M., et la chaîne Benjamin. «La séquence de séquenceurs: l'histoire de l'ADN de séquençage.”Génomique. Academic Press, Jan. 2016. la toile. 30 mars. 2017
3.Pareek, Chandra Shekhar, Rafal Smoczynski et Andrzej Tretyn. «Les technologies de séquençage et le séquençage du génome.»Journal of Applied Genetics. Springer-Verlag, nov. 2011. la toile. 30 mars. 2017
Image gracieuseté:
1. «MAXAM GILBERT SEQUENCING» à plusieurs reprises à English Wikipedia (CC par 3.0) via Commons Wikimedia
2. «Sanger-séquençage» par Estevezj - propre travail (CC BY-SA 3.0) via Commons Wikimedia