Différence entre NGS et Sanger Sequencing
Différence clé - NGS vs Sanger Séquençage
Le séquençage de prochaine génération (NGS) et le séquençage Sanger sont deux types de techniques de séquençage nucléotidique développées au fil du temps. La méthode de séquençage Sanger a été largement utilisée pendant de nombreuses années et NGS l'a remplacé récemment en raison de ses avantages. La principale différence entre NGS et le séquençage Sanger est que NGS works on the principle of sequencing millions of sequences simultaneously in a rapid way through a sequencing system while the Sanger Sequencing works on the principal of chain termination due to selective incorporation of dideoxynucleotides by DNA polymerase enzyme during the DNA replication and resulting fragment separation by capillary électrophorèse.
CONTENU
1. Aperçu et différence clé
2. Qu'est-ce que le séquençage nucléotidique
3. Qu'est-ce que NGS
4. Qu'est-ce que le séquençage Sanger
5. Comparaison côte à côte - Séquençage NGS vs Sanger
6. Résumé
Qu'est-ce que le séquençage nucléotidique?
Les informations génétiques sont stockées dans les séquences nucléotidiques de l'ADN ou de l'ARN d'un organisme. Le processus de détermination de l'ordre correct des nucléotides (en utilisant quatre bases) dans un fragment donné (dans un gène, une grappe de gènes, du chromosome et du génome complet) est appelé séquençage nucléotidique. Il est très important dans les études génomiques, les études médico-légales, la virologie, la systématique biologique, le diagnostic médical, la biotechnologie et dans de nombreux autres domaines pour analyser la structure et la fonction des gènes. Il existe différents types de méthodes de séquençage développées par les scientifiques. Parmi eux, Séquençage Sanger développé par Frederick Sanger en 1977 a été largement utilisé et popularisé pendant une longue période jusqu'à Séquençage de prochaine génération remplacé.
Qu'est-ce que NGS?
Le séquençage de prochaine génération (NGS) est un terme utilisé pour désigner les processus de séquençage moderne à haut débit. Il décrit un certain nombre de technologies de séquençage modernes différentes qui ont révolutionné les études génomiques et la biologie moléculaire. Ces techniques sont le séquençage Illumina, le séquençage de Roche 454, le séquençage de protons ion et le séquençage solide (séquençage par détection de ligature oligo). Les systèmes NGS sont plus rapides et moins chers. Quatre méthodes principales de séquençage d'ADN sont utilisées dans les systèmes NGS, à savoir; pyroséquençage, séquençage par synthèse, séquençage par ligature et séquençage de semi-conducteur ion. Un grand nombre de volets d'ADN ou d'ARN (millions de) peuvent être séquencés parallelly. Il permet le séquençage de l'ensemble du génome des organismes dans une courte période, contrairement au séquençage de Sanger qui prend plus de temps.
NGS présente de nombreux avantages par rapport à la méthode Sanger de séquençage conventionnel. Il s'agit d'un processus à grande vitesse, plus précis et rentable qui peut être effectué avec un petit échantillon. Les NG peuvent être utilisés dans les études métagénomiques, dans la détection des variations au sein d'un génome individuel en raison des insertions et des suppressions, etc. et dans l'analyse des expressions génétiques.
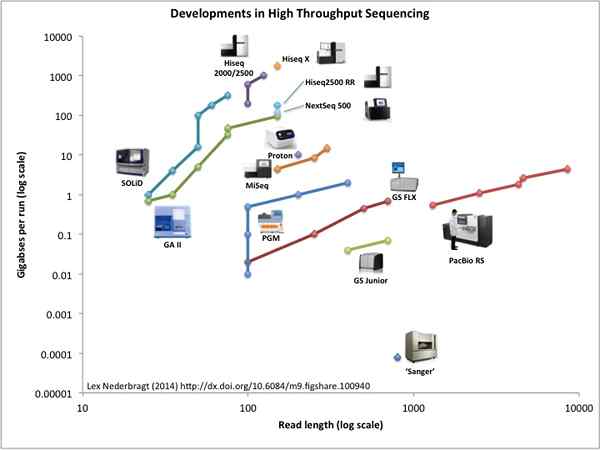
Figure_1: Développements dans le séquençage NGS
Qu'est-ce que le séquençage Sanger?
Le séquençage de Sanger est une méthode de séquençage développée par Frederick Sanger et ses collègues en 1977 pour déterminer l'ordre nucléotidique précis d'un fragment d'ADN donné. Il est également connu sous le nom séquençage de terminaison de la chaîne ou Séquençage de la didésoxy. Le principe de travail de cette méthode est la terminaison de la synthèse des brins par l'incorporation sélective d'une chaîne terminant les didésoxynucléotides (DDNTP) tels que DDGTP, DDCTP, DDATP et DDTTP par l'ADN polymérase lors de la réplication de l'ADN. Les nucléotides normaux ont 3 groupes OH pour la formation d'une liaison phosphodiester entre les nucléotides adjacents pour poursuivre la formation du brin. Cependant, les DDNTP n'ont pas ce groupe OH 3 'et ne sont pas en mesure de former des liaisons phosphodiester entre les nucléotides. Par conséquent, l'allongement de la chaîne est cessé.
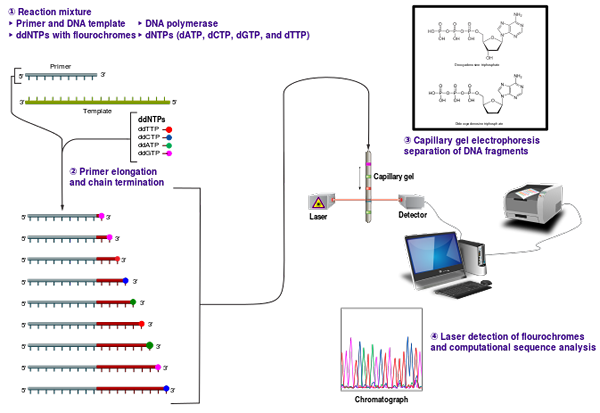
Dans cette méthode, l'ADN simple brin à séquencé sert de brin de modèle pour in vitro Synthèse d'ADN. D'autres exigences sont l'amorce oligonucléotide, les précurseurs désoxynucléotidiques et l'enzyme d'ADN polymérase. Lorsque les extrémités flanquantes du fragment cible sont connues, les amorces peuvent être facilement conçues pour la réplication de l'ADN. Quatre réactions de synthèse d'ADN distinctes sont effectuées dans quatre tubes séparés. Chaque tube a des DDNTP séparés, avec d'autres exigences. À partir du nucléotide particulier, un mélange de DNTP et de DDNTP est ajouté. De même, quatre réactions distinctes sont effectuées en quatre tubes avec quatre mélanges. Après les réactions, la détection des fragments d'ADN et la conversion du modèle de fragment en informations de séquence sont effectuées. Les fragments d'ADN résultants sont dénaturés à la chaleur et séparés par électrophorèse sur gel. Si des nucléotides radioactifs sont utilisés, le modèle de bandes dans le gel de polyacrylamide peut être visualisé par autoradiographie. Lorsque cette méthode utilise les didésoxynucléotides marqués par fluorescence, il peut être atténué dans la lecture du gel et passé à travers un faisceau de laser pour être détecté par le détecteur fluorescent. Pour éviter les erreurs qui pourraient survenir lorsqu'une séquence est lue par l'œil et entrer manuellement dans un ordinateur, cette méthode s'est développée dans l'utilisation d'un séquenceur automatisé couplé à l'ordinateur.
Ceci est la méthode utilisée pour séquencer l'ADN du projet du génome humain. Cette méthode est toujours utilisée avec des modifications avancées car elle donne des informations de séquence précises bien qu'ils soient un processus coûteux et lent.

Figure_2: séquençage Sanger
Quelle est la différence entre le séquençage NGS et Sanger?
Séquençage NGS vs Sanger | |
| Le séquençage de prochaine génération (NGS) se réfère aux processus de séquençage moderne à haut débit. Il décrit un certain nombre de technologies de séquençage modernes différentes | Le séquençage Sanger est une méthode de séquençage développée par Frederick Sanger pour déterminer l'ordre nucléotidique précis d'un fragment d'ADN donné. |
| Rentabilité | |
| NGS est un processus moins cher car il réduit le temps, la puissance de l'homme et les produits chimiques. | C'est un processus coûteux car il faut du temps, de la puissance de l'homme et plus de produits chimiques. |
| Vitesse | |
| Ceci est plus rapide car la détection chimique et la détection de signal de nombreux brins se produisent parallèles. | Cela prend du temps puisque la détection des produits chimiques et la détection du signal se produisent sous forme de deux processus distincts et que ce n'est que sur le brin peut lire à la fois. |
| Fiabilité | |
| NGS est fiable. | Le séquençage Sanger est moins fiable |
| Taille de l'échantillon | |
| NGS nécessite moins d'ADN. | Cette méthode a besoin d'une grande quantité de modèle ADN. |
| Bases d'ADN par fragment séquencé | |
| Le nombre de bases d'ADN par fragment séquencé est inférieure à la méthode Sanger | Les séquences de génération sont plus longues que les séquences NGS. |
Résumé - NGS vs Sanger Séquençage
NGS et le séquençage Sanger sont des techniques de séquençage nucléotidique largement utilisées en biologie moléculaire. Le séquençage Sanger est une méthode de séquençage précoce qui a été remplacée par NGS. La principale différence entre NGS et le séquençage Sanger est que NGS est un processus à grande vitesse, plus précis et rentable que le séquençage Sanger. Les deux techniques ont créé des épidémies majeures en génétique et en biotechnologie.
Référence:
1. Nowrousian, Minou. «Techniques de séquençage de nouvelle génération pour les micro-organismes eucaryotes: solutions basées sur le séquençage aux problèmes biologiques." Cellule eukaryotique. American Society for Microbiology, sept. 2010. la toile. 18 février. 2017
2. Sanger, F., S. Nicklen, et un. R. Coulson. «Séquençage d'ADN avec des inhibiteurs de la chaîne.»Actes de l'Académie nationale des sciences 74.12 (1977): 5463-467. la toile.
3. Liu, Lin, Yinhu Li, Siliang Li, Ni Hu, Yimin He, Ray Pong, Danni Lin, Lihua Lu et Maggie Law. «Comparaison des systèmes de séquençage de nouvelle génération.»Journal of Biomedicine and Biotechnology 2012 (2012): 1-11. la toile.
Image gracieuseté:
«Sanger-séquençage» par Estevezj - propre travail (CC BY-SA 3.0) via Commons Wikimedia
«Développements dans le séquençage de prochaine génération» de Nederbragt, Lex (2012) - (CC par 3.0) via Commons Wikimedia