Différence entre la réparation des mésappariements et la réparation d'excision des nucléotides
Différence clé - Réparation de mésappariement vs réparation d'excision nucléotidique
Des dizaines et des milliers de dommages à l'ADN se produisent dans la cellule par jour. Il induit des modifications aux processus cellulaires tels que la réplication, la transcription ainsi que la viabilité de la cellule. Dans certains cas, les mutations causées par ces dommages à l'ADN peuvent entraîner des maladies délétères comme les cancers et les syndromes associés au vieillissement (Ex: Progeria). Indépendamment de ces dommages, la cellule initie un mécanisme de réparation en cascade hautement organisé appelé réponses aux dommages à l'ADN. Plusieurs systèmes de réparation d'ADN ont été identifiés dans le système cellulaire; Ceux-ci sont connus sous le nom de réparation d'excision de base (BER), réparation de décalage (MMR), réparation d'excision nucléotidique (NER), réparation de rupture à double brin. La réparation de l'excision nucléotidique est un système très polyvalent qui reconnaît les lésions d'ADN de distorsion hélicoïdale volumineuses et les supprime. D'un autre côté, la réparation de l'inadéquation remplace les bases mal en société pendant la réplication. La principale différence entre la réparation des mésappariements et la réparation d'excision des nucléotides est que La réparation d'excision des nucléotides (NER) est utilisée pour éliminer les dimères de pyrimidine formées par l'irradiation UV et les lésions hélicoïdales volumineuses causées par les adduits chimiques tandis. En plus des bases maltraitées, les protéines du système MMR peuvent également réparer les boucles d'insertions / délétions (IDL) qui sont les résultats du glissement de la polymérase pendant la réplication de séquences d'ADN répétitives.
CONTENU
1. Aperçu et différence clé
2. Qu'est-ce que la réparation de décalage
3. Qu'est-ce que la réparation d'excision nucléotidique
4. Comparaison côte à côte - Réparation de mésappariement vs réparation d'excision nucléotidique
5. Résumé
Qu'est-ce que la réparation d'excision nucléotidique?
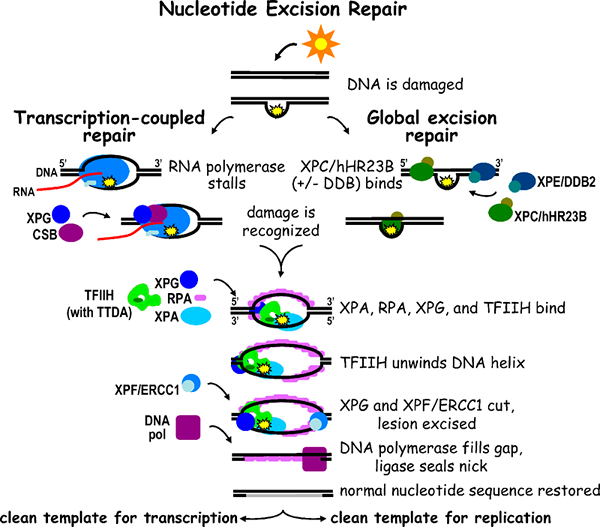
La caractéristique la plus distinguée de la réparation d'excision nucléotidique est qu'il répare les dommages nucléotidiques modifiés causés par des distorsions significatives dans l'ADN double hélice. Il est observé dans presque tous les organismes qui ont été examinés à jour. UVR A, UVR B, UVR C (Exinucléases) UVR D (A Hélicase) sont les enzymes les plus connues impliquées dans le NER qui déclenchent la réparation de l'ADN dans l'organisme modèle Ecoli. UVR ABC Le complexe enzymatique Multi-Subunités produit les polypeptides UVR A, UVR B, UVR C. Les gènes codés pour les polypeptides susmentionnés sont UVR A, UVR B, UVR C. Les enzymes UVR A et B reconnaissent collectivement la distorsion induite par les dommages causée à la double hélice d'ADN tel que les gradateurs de pyrimidine en raison de l'irradiation UV. UVR A est une enzyme ATPase et c'est une réaction autocatalytique. Ensuite, UVR A quitte l'ADN tandis que le complexe UVR BC (nucléase actif) clignent l'ADN des deux côtés des dommages qui ont catalysé par l'ATP. Une autre protéine appelée UVR D codée par le gène UVRD est une enzyme d'hélicase II dénouille l'ADN qui résulte de la libération du segment d'ADN endommagé simple brin. Cela laisse une lacune dans l'hélice d'ADN. Après l'excision du segment endommagé, un écart de nucléotide de 12-13 reste dans le brin d'ADN. Ceci est rempli par l'enzyme ADN polymérase I et le Nick est scellé par l'ADN ligase. L'ATP est requis à trois étapes de cette réaction. Le mécanisme NER peut également être identifié chez les humains de type mammifère. Chez l'homme, l'état de la peau appelée Xeroderma pigmentosum est due aux dimères d'ADN causés par l'irradiation UV. Les gènes XPA, XPB, XPC, XPD, XPE, XPF et XPG produisent des protéines pour remplacer les dommages à l'ADN. Les protéines des gènes XPA, XPC, XPE, XPF et XPG ont l'activité de nucléase. D'un autre côté, les protéines des gènes XPB et XPD montrent l'activité hélicase qui analogue à UVR D E coli.

Figure 01: Réparation d'excision nucléotidique
Qu'est-ce que la réparation de décalage?
Le système de réparation de décalage est initié pendant la synthèse de l'ADN. Même avec la sous-unité € fonctionnelle, l'ADN polymérase III permet l'incorporation d'un mauvais nucléotide pour la synthèse tous les 108 paires de bases. Les protéines de réparation des mésappariements reconnaissent ce nucléotide, l'accise et le remplacent par le nucléotide correct responsable du degré final de précision. La méthylation de l'ADN est essentielle pour les protéines MMR pour reconnaître le brin parent du brin nouvellement synthétisé. La méthylation du nucléotide d'adénine (A) dans un motif GATC d'un brin nouvellement synthétisé est un peu retardé. D'un autre côté, le nucléotide d'adénine du brin parental dans le motif GATC a déjà méthylé. Les protéines MMR reconnaissent le brin nouvellement synthétisé par cette différence par rapport au brin parent et commencent la réparation de la décalage dans un brin nouvellement synthétisé avant qu'il ne soit méthylé. Les protéines MMR dirigent leur activité de réparation pour acciser le mauvais nucléotide avant que le brin d'ADN nouvellement répliqué ne soit méthylé. Les enzymes Mut H, Mut L et Mut S codées par des gènes Mut H, Mut L, Mut S catalysent ces réactions en Ecoli. La protéine Mut S reconnaît sept des huit paires de bases de décalage possibles à l'exception de C: C, et se lie au site de décalage dans l'ADN duplex. Avec des ATP liés, Mut L et Mut S rejoignent le complexe plus tard. Le complexe transloque à quelques milliers de paires de bases jusqu'à ce qu'il trouve un motif GATC hémiméthylé. L'activité de nucléase dormante de la protéine mut h est activée une fois qu'elle trouve un motif GATC hémiméthylé. Il clive le brin d'ADN non méthylé, laissant un nick de 5 'à G nucléotidique du motif GATC non méthylé (brin d'ADN nouvellement synthétisé). Ensuite, le même brin de l'autre côté de l'inadéquation est entaillé par mut h. Dans le reste des étapes, les actions collectives de la protéine d'hélicase UVR D, de l'hélicase, du Mut U, du SSB et de l'exonucléase I, accisent le nucléotide incorrect dans l'ADN simple brin. L'écart qui est formé dans l'excision est comblé par l'ADN polymérase III et scellé par ligase. Un système similaire peut être identifié chez les souris et les humains. La mutation de HMLH1, HMSH1 et HMSH2 humain est impliquée dans un cancer du côlon héréditaire non-polypose qui désacule la division cellulaire des cellules du côlon.
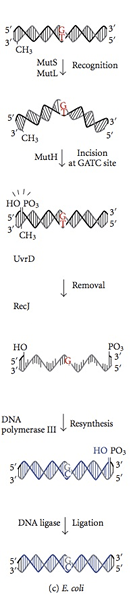
Figure 02: Réparation de décalage
Quelle est la différence entre la réparation de l'inadéquation et la réparation d'excision des nucléotides?
Réparation de mésappariement vs réparation d'excision nucléotidique | |
| Le système de réparation de mésapparie se produit pendant la post-réplication. | Ceci est impliqué dans l'élimination des dimères de pyrimidine due à u.V Irradiation et autres lésions d'ADN en raison de l'addition chimique. |
| Enzymes | |
| Il est catalysé par Mut S, Mut L, Mut H, UVR D, SSB et exonucléase I. | Il est catalysé par UVR A, UVR B, UVR C, enzymes UVRD. |
| Méthylation | |
| Il est essentiel d'initier la réaction. | La méthylation de l'ADN n'est pas nécessaire pour lancer la réaction. |
| Action des enzymes | |
| Mut H est une endonucléase. | UVR B et UVR C sont des exonucléases. |
| Occasion | |
| Cela se produit spécifiquement pendant la réplication. | Cela se produit lorsqu'il est exposé à vous.V ou mutagènes chimiques, pas pendant la réplication |
| Conservation | |
| C'est hautement conservé | Ce n'est pas hautement conservé. |
| Combler les lacunes | |
| Il se fait par l'ADN polymérase III. | Il se fait par l'ADN polymérase I. |
Résumé - Réparation de mésappariement vs réparation d'excision nucléotidique
La réparation des mésappariements (MMR) et la réparation d'excision des nucléotides (NER) sont deux mécanismes qui ont lieu dans la cellule afin de rectifier les dommages et les distorsions de l'ADN qui sont causées par divers agents. Ceux-ci sont collectivement nommés comme des mécanismes de réparation de l'ADN. La réparation de l'excision nucléotidique répare les dommages nucléotidiques modifiés, généralement ces dommages importants de la double hélice d'ADN qui se produisent en raison de l'exposition à u.V Irradiation et adduits chimiques. Les protéines de réparation des mésappariements reconnaissent le mauvais nucléotide, l'accise et le remplacent par un nucléotide correct. Ce processus est responsable du degré final de précision pendant la réplication.
Référence:
1.Cooper, Geoffrey M. «Réparation de l'ADN."La cellule: une approche moléculaire. 2e édition.U.S. Bibliothèque nationale de médecine, 01 janvier. 1970. la toile. 09 mars. 2017.
2.»Mécanismes et fonctions de réparation de décalage de l'ADN.»Recherche cellulaire. U.S. Bibliothèque nationale de médecine, n.d. la toile. 09 mars. 2017.
Image gracieuseté:
1. «Journal d'excision nucléotidique.PBIO.0040203.G001 ”par Jill O. Fuss, Priscilla k. Cooper - (CC par 2.5) Via Commons Wikimedia
2. «ADN Cismatch Repair Ecoli» par Kenji Fukui - (CC par 4.0) via Commons Wikimedia