Différence entre les puces à ADN et le séquençage de prochaine génération

Différence clé - ADN ADN VS Sequençage de prochaine génération
Les processus de séquençage de l'ADN sont largement utilisés dans les domaines de la biotechnologie, de la virologie, du diagnostic médical et des sciences judiciaires. C'est un processus qui détermine l'ordre exact des nucléotides, adénine, guanine, thymine et cytosine présents dans une molécule d'ADN. Les procédures de séquençage ADN sont devenues un accélérateur pour les découvertes miraculeuses dans la recherche médicale et biologique. Ces méthodes de séquençage ont évolué vers le séquençage d'un génome complet d'organismes individuels, y compris les humains et d'autres espèces vivantes. Les microréseaux et le séquençage de prochaine génération sont des procédures de séquençage d'ADN modernes. La technique de puces à ADN est spécifiquement basée sur l'hybridation qui contient un ensemble de cibles connues. Le séquençage de prochaine génération est basé sur la synthèse (qui utilise l'ADN polymérase pour incorporer des nucléotides) et a la capacité de séquencer le génome entier indépendant des cibles sélectionnées précédemment. C'est la principale différence entre les puces à ADN et le séquençage de prochaine génération.
CONTENU
1. Aperçu et différence clé
2. Qu'est-ce que les microréseaux
3. Qu'est-ce que le séquençage de prochaine génération
4. Similitudes entre les microréseaux et le séquençage de prochaine génération
5. Comparaison côte à côte - Aicroréseau vs séquençage de prochaine génération sous forme tabulaire
6. Résumé
Qu'est-ce que les microréseaux?
ADN ADN est utilisé comme un outil de laboratoire afin d'identifier des milliers d'expressions de gènes différentes en même temps. C'est une surface solide, je.e., diapositive de microscope, qui contient une collection de taches d'ADN microscopiques imprimées dessus. Chaque tache imprimée contient une séquence de gènes connue ou un gène. Ces sondes connues imprimées sur la diapositive servent de sondes afin de détecter l'expression des gènes. Ceci est connu comme un transcriptome. L'hybridation entre deux brins d'ADN est le principal principe que les microréseaux sont basés sur. Il s'agit de l'appariement de base complémentaire des séquences d'acide nucléique avec la formation de liaisons hydrogène.

Figure 01: ADN
Initialement, les molécules d'ARNm sont collectées à partir de l'échantillon expérimental et de l'échantillon de référence obtenu à partir d'un individu sain. Des échantillons expérimentaux sont obtenus auprès d'individus malades; Par exemple, un individu souffrant de cancer. Une fois obtenus, les deux échantillons d'ARNm sont convertis en ADNc (ADN complémentaire). Ensuite, chaque échantillon est étiqueté à l'aide d'une sonde fluorescente. Les sondes fluorescentes sont de différentes couleurs pour distinguer l'ADNc de l'échantillon de l'ADNc de référence. Afin d'initier la liaison des molécules d'ADNc à la diapositive de puces à ADN, les deux échantillons sont mélangés ensemble. L'hybridation est le processus par lequel les molécules d'ADNc s'attachent aux sondes d'ADN sur la diapositive de puces à ADN. Une fois l'hybridation terminée, une série de réactions a lieu afin d'identifier et de mesurer l'expression de chaque gène avec l'apparition de différentes couleurs en fonction de la quantité du gène exprimé. Les résultats des puces à ADN sont utilisées dans la création d'un profil d'expression génique qui peut être utilisé pour identifier différentes maladies.
Qu'est-ce que le séquençage de prochaine génération?
Le séquençage de prochaine génération (NGS) est une méthode avancée de séquençage génétique. Son principe est similaire à celui du séquençage Sanger, qui dépend de l'électrophorèse capillaire. Dans NGS, le brin génomique est fragmenté et ligaturé en un brin de modèle. Les bases de chaque brin sont identifiées par les signaux émis au cours de son processus de ligature. Dans la méthode de séquençage Sanger, trois étapes distinctes, séquençage, séparation et détection sont impliquées. En raison de ces étapes distinctes, l'automatisation de la préparation des échantillons est limitée dans le débit. Dans NGS, la technique est développée en utilisant le séquençage basé sur le tableau avec la combinaison des étapes de la procédure de séquençage Sanger qui peut entraîner des millions de séries de réaction parallèles en même temps; il en résulte une vitesse et un débit élevé.
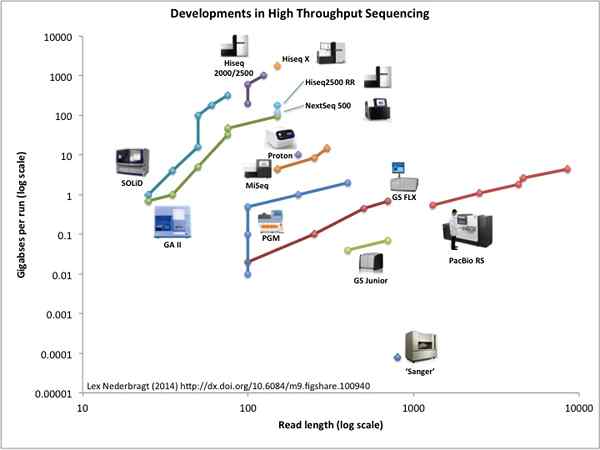
Figure 02: Développements dans NGS
NGS est composé de trois étapes; Préparation de la bibliothèque (création de bibliothèques à l'aide d'une fragmentation aléatoire de l'ADN), d'amplification (amplification de la bibliothèque en utilisant l'amplification clonale et la PCR) et le séquençage. Les processus de séquençage du génome qui sont effectués pour des durées extrêmement longs à l'aide de la procédure de séquençage Sanger pourraient être achevés en quelques heures en utilisant NGS.
Quelle est la similitude entre les microréseaux et le séquençage de prochaine génération?
- Les microréseaux et le séquençage de prochaine génération sont développés en utilisant le séquençage basé sur le tableau.
Quelle est la différence entre les puces à ADN et le séquençage de prochaine génération?
ADNI | |
| Microarray est une collection de taches d'ADN microscopiques attachées à une surface solide, qui est utilisée pour mesurer les niveaux d'expression de grand nombre de gènes simultanément. | NGS (séquençage de nouvelle génération) est une technologie de séquençage d'ADN à haut débit non basée sur les sanger qui facilite des millions ou des milliards de brins d'ADN à séquenter en parallèle. |
| Interactions avec l'antigène | |
| Les microréseaux sont basés sur l'hybridation composée d'un ensemble de cibles connues. | NGS est basé sur la synthèse qui utilise l'ADN polymérase pour incorporer des nucléotides et est indépendant des cibles précédemment sélectionnées. |
Résumé - ADN ADN VS Sequençage de prochaine génération
Dans le contexte de la recherche, le séquençage de l'ADN est devenu un accélérateur important. Il est largement utilisé en biotechnologie, diagnostic médical et études médico-légales. Il a évolué et développé en procédures de séquençage plus efficaces et rapides. Les microréseaux et les NG sont deux techniques avancées de séquençage d'ADN présent. Les deux sont développés en utilisant le séquençage basé sur le tableau. La technique de microréseau repose sur l'hybridation tandis que le NGS est basé sur la synthèse, qui utilise l'ADN polymérase pour incorporer les nucléotides. C'est la principale différence entre les puces à ADN et le séquençage de prochaine génération.
Télécharger la version PDF de ADN Microarray vs Sequençage de Next Generation
Vous pouvez télécharger la version PDF de cet article et l'utiliser à des fins hors ligne selon la note de citation. Veuillez télécharger la version PDF ici différence entre ADN ADN et séquençage de prochaine génération
Les références:
1.Behjati, Sam et Patrick S Tarpey. «Qu'est-ce que le séquençage de prochaine génération?"Archives de maladies infantiles. Édition d'éducation et de pratique, BMJ Publishing Group, DEC. 2013, disponible ici. Consulté le 23 août. 2017
2.Bumgarner, Roger. «Aperçu des puces à ADN.»Protocoles actuels en biologie moléculaire, John Wiley et Sons Inc., 9 février. 2016, disponible ici. Consulté le 23 août. 2017.
3.«Technologie ADN à ADN.»National Human Genome Research Institute (NHGRI), disponible ici. Consulté le 23 août. 2017.
Image gracieuseté:
1. «ADN microarray» de Guillaume Paumier (utilisateur: Guillom) - Propre travaux (CC BY-SA 3.0) via Commons Wikimedia
2. «Développements dans le séquençage de prochaine génération» de Nederbragt, Lex (2012) - (CC par 3.0) via Commons Wikimedia