Différence entre l'homologie et la similitude en bioinformatique
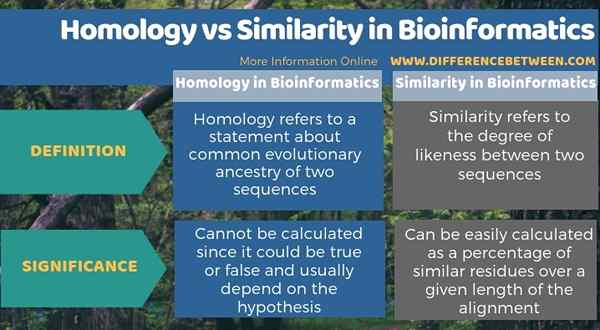
Le différence clé entre l'homologie et la similitude en bioinformatique L'homologie fait référence à une déclaration sur l'ascendance évolutive commune de deux séquences tandis que la similitude se réfère au degré de ressemblance entre deux séquences.
La bioinformatique est un domaine scientifique qui combine la biologie, l'ingénierie de l'information, l'informatique, les mathématiques et les statistiques pour analyser et interpréter les données biologiques. L'homologie et la similitude sont deux termes que nous utilisons dans le domaine de la bioinformatique. Nous pouvons facilement calculer la similitude en pourcentage de résidus similaires sur une longueur donnée de l'alignement. Cependant, nous ne pouvons pas calculer l'homologie car elle pourrait être vraie ou fausse et dépendre généralement de l'hypothèse utilisée.
CONTENU
1. Aperçu et différence clé
2. Qu'est-ce que l'homologie en bioinformatique
3. Quelle est la similitude en bioinformatique
4. Similitudes entre l'homologie et la similitude en bioinformatique
5. Comparaison côte à côte - Homologie vs similitude en bioinformatique sous forme tabulaire
6. Résumé
Qu'est-ce que l'homologie en bioinformatique?
L'homologie en bioinformatique fait référence à l'homologie biologique entre l'ADN, l'ARN et les séquences de protéines qui sont définies en termes de propriétés ancestrales partagées dans l'arbre évolutif de la vie. En d'autres termes, c'est l'ascendance évolutive commune de deux séquences. La raison d'une telle occurrence pourrait être due à des événements de spéciation (orthologues), à des événements de transfert de gènes horizontaux (xénologues) ou à des événements de duplication (paralogues).

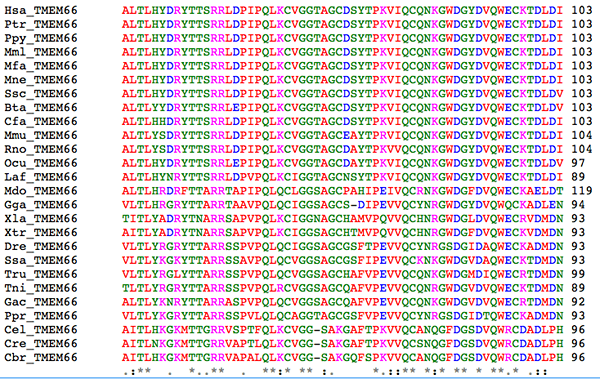
Figure 01: Alignement de la séquence multiple
Il est possible de déduire l'homologie entre l'ADN, l'ARN ou les protéines par leur similitude d'acide aminé ou de séquence nucléotidique. Une similitude significative sert de propriété de preuve forte pour déduire que deux séquences sont liées à une séquence ancestrale commune avec des changements évolutifs. Les alignements de plusieurs séquences indiquent les régions de chaque séquence de nature homologue.
Quelle est la similitude en bioinformatique?
En bioinformatique, la similitude évalue la similitude entre deux protéines ou séquences nucléotidiques. Il y a deux étapes principales vers ce processus. L'étape initiale est l'alignement par paire, qui aide à trouver l'alignement optimal entre deux séquences (y compris les lacunes) en utilisant des algorithmes tels que Blast, Fasta et Lalign. Après l'alignement par paire, il est nécessaire d'obtenir deux paramètres quantitatifs à partir de chaque comparaison par paire. Ils sont l'identité et la similitude. Dans l'explosion, les similitudes de recherche sont connues sous le nom de positives.

Figure 02: Alignement par paire
Une mutation conservatrice se produit lorsqu'un acide aminé mute à un résidu similaire tout en préservant les propriétés physiochimiques. Par exemple, si l'arginine mute en lysine avec une charge positive +1, elle est acceptée car les deux acides aminés sont similaires en propriété et ne changent pas la protéine traduite. Par conséquent, les mesures de similitude dépendent des critères de la façon dont deux résidus d'acides aminés sont les uns aux autres.
Quelles sont les similitudes entre l'homologie et la similitude en bioinformatique?
- L'homologie et la similitude sont deux termes techniques que nous rencontrons en bioinformatique.
- De plus, les deux termes se réfèrent à l'analyse moléculaire des séquences.
Quelle est la différence entre l'homologie et la similitude en bioinformatique?
L'homologie fait référence à une déclaration sur l'ascendance évolutive commune de deux séquences tandis que la similitude se réfère au degré de ressemblance entre deux séquences. C'est donc la principale différence entre l'homologie et la similitude en bioinformatique. De plus, l'homologie ne peut pas être calculée car elle pourrait être vraie ou fausse et dépendre généralement de l'hypothèse tandis que la similitude peut être facilement calculée en pourcentage de résidus similaires sur une longueur donnée de l'alignement. Par conséquent, c'est une différence significative entre l'homologie et la similitude en bioinformatique.
L'infographie suivante énumère la différence entre l'homologie et la similitude en bioinformatique.
Résumé - Homologie vs similitude en bioinformatique
En bref, la principale différence entre l'homologie et la similitude en bioinformatique réside dans leurs définitions. L'homologie est une déclaration d'ascendance évolutive commune de deux séquences tandis que la similitude est la ressemblance entre deux séquences. L'homologie se produit en raison des orthologues, des paralogues et des xénologues. Lors de la déduction de la similitude, il est possible d'utiliser des algorithmes tels que Fasta, Blast et Lalign. L'homologie ne peut pas être exprimée comme un calcul, mais la similitude pourrait être exprimée en pourcentage de résidus similaires sur une longueur donnée de l'alignement. Ainsi, c'est le résumé de la différence entre l'homologie et la similitude en bioinformatique.
Référence:
1.«Homologue.”Homologue - Bioinformatique.Org wiki, disponible ici.
2. «Identité et similitude - une mesure quantitative.«Identité et similitude - une mesure quantitative, disponible ici.
Image gracieuseté:
1. «Un extrait d'un alignement de séquences multiples des protéines TMEM66» par WIKID25 - Clustalw open source et séquences de la base de données publique de protéines NCBI (CC0) via Commons Wikimedia
2. «Alignement mondial par paire» par Alfat003 - Propre travaux (CC BY-SA 4.0) via Commons Wikimedia