Différence entre Blast et Fasta
Le différence clé entre Blast et Fasta est que Le Blast est un outil d'alignement de base disponible sur le site Web du National Center for Biotechnology Information tandis que Fasta est un outil de recherche de similitude disponible sur le site Web de l'Institut européen Bioinformatics Institute.
Blast et Fasta sont deux logiciels largement utilisés pour comparer les séquences biologiques d'ADN, des acides aminés, des protéines et des nucléotides de différentes espèces et recherchent leurs similitudes. Ces algorithmes ont été écrits en gardant la vitesse à l'esprit. Parce que, lorsque les scientifiques ont pu isoler l'ADN en laboratoire au milieu des années 80, cela a soulevé la nécessité de comparer et de trouver des gènes identiques pour des recherches supplémentaires à grande vitesse. Par conséquent, ces deux logiciels ont été développés d'une manière que l'utilisateur peut faire une recherche rapide de séquences similaires avec celle de leurs séquences de requête.
Blast est un acronyme pour Outil de recherche d'alignement local de base et utilise l'approche localisée pour comparer les deux séquences. Fasta est un logiciel faisant référence à Fast A où A représente tous. Ici, le logiciel fonctionne avec des alphabets comme Fast A pour le séquençage d'ADN et P pour les protéines. Blast et Fasta sont très rapides pour comparer n'importe quelle base de données du génome et sont donc très viables sur le plan monétaire ainsi qu'à gagner du temps.
CONTENU
1. Aperçu et différence clé
2. Qu'est-ce que Blast
3. Qu'est-ce que Fasta
4. Similitudes entre Blast et Fasta
5. Comparaison côte à côte - explosion vs fasta sous forme tabulaire
6. Résumé
Qu'est-ce que Blast?
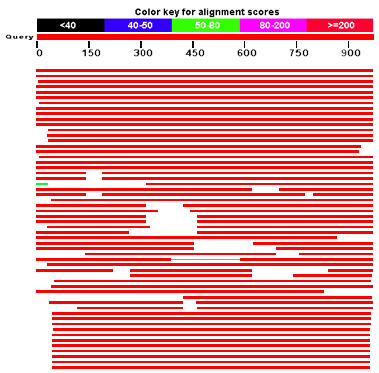
BLAST est l'un des logiciels de bioinformatique les plus utilisés qui ont été développés en 1990. Depuis lors, il est disponible pour tout le monde sur le site NCBI. De plus, toute personne peut accéder à ce logiciel et l'utiliser. De plus, BLAST est un logiciel qui a besoin de données d'entrée ou de séquences au format Fasta. Mais, il donne des données de sortie au format de texte brut, HTML ou XML. BLAST travaille sur un principe de recherche de similitudes localisées entre deux séquences et la liste courte les séquences similaires, et après cela, il recherche des similitudes de quartier.

Figure 01: Résultats de l'explosion
Ainsi, ce logiciel recherche un nombre élevé de régions locales similaires et donne le résultat sur l'atteinte d'une valeur de seuil. Cependant, ce processus diffère du logiciel précédent, qui recherche d'abord toute la séquence, puis fait la comparaison, et a donc pris beaucoup de temps.
Outre la vérification de la similitude ci-dessus, Blast a de nombreuses autres utilisations telles que la cartographie de l'ADN, en comparant deux gènes identiques dans différentes espèces, créant un arbre phylogénétique, etc.
Qu'est-ce que Fasta?
Fasta est un logiciel d'alignement de séquence protéique. David J. Lipman et William R. Pearson a décrit ce logiciel en 1985. Bien que l'utilisation initiale de ce logiciel consistait à comparer uniquement les séquences de protéines, la version modifiée de celui-ci a également pu comparer les séquences d'ADN. Ici, ce logiciel utilise le principe de trouver la similitude entre deux séquences statistiquement. Il correspond à une séquence de l'ADN ou de la protéine avec une autre séquence par méthode d'alignement de séquence locale.

Figure 02: Fasta
Cependant, il recherche des régions locales pour la similitude, mais pas la meilleure correspondance entre deux séquences. Puisque ce logiciel compare parfois des similitudes localisées, il peut également proposer des décalages. Dans une séquence, Fasta prend une petite partie connue sous le nom de k-tuples, où les tuples peuvent être de 1 à 6 et correspond aux tuples k de l'autre séquence. À la fin du processus d'appariement, lorsqu'il atteint une valeur de seuil, il produit le résultat.
Quelles sont les similitudes entre Blast et Fasta?
- Blast et Fasta sont des outils de bioinformatique utilisés pour comparer les séquences de protéines et d'ADN pour les similitudes.
- De plus, les deux programmes utilisent une stratégie de notation pour faire des comparaisons entre les séquences.
- De plus, les deux outils produisent des résultats très précis.
Quelle est la différence entre Blast et Fasta?
Blast est un outil pour vérifier la similitude entre les séquences biologiques. D'un autre côté, Fasta est un autre programme qui facilite la vérification de la similitude des séquences de protéines et d'ADN. Cependant, par rapport à Fasta, le logiciel BLAST est très populaire car il produit des résultats plus précis et rapides. Par conséquent, c'est la différence entre Blast et Fasta. De plus, contrairement à Fasta, le programme BLAST est modifiable en fonction de la nécessité de l'utilisateur. Par conséquent, c'est une autre différence entre Blast et Fasta.
L'infographie ci-dessous sur la différence entre Blast et Fasta fournit plus de détails.

Résumé - BLAST VS FASTA
Blast et Fasta sont deux programmes qui permettent à l'utilisateur de comparer sa séquence de requête avec les séquences des bases de données existantes et de vérifier les similitudes. Le but initial de Fasta était de comparer uniquement les séquences de protéines. Mais, la version modifiée de ce logiciel facilite les comparaisons de séquences protéiques et ADN. Bien que Fasta soit un bon logiciel, la plupart des gens utilisent l'outil d'alignement BLAST car il est plus populaire et produit des résultats plus précis et rapides que Fasta. De plus, le souffle est modifiable en fonction des exigences de l'utilisateur. En bref, cela résume la différence entre Blast et Fasta.
Référence:
1.Madden, Thomas. «L'outil d'analyse de séquence de souffle.»Rapports actuels de neurologie et de neurosciences., U.S. Bibliothèque nationale de médecine, 15 mars. 2013. Disponible ici
Image gracieuseté:
1.«CCDC132 Blast Results» par NCBI BLAST - NCBI BLAST, (Domaine public) via Commons Wikimedia
2.«Région promotrice de FAM149A (Fasta Format)» par Larsongcd - Propre travaux, (CC BY-SA 3.0) via Commons Wikimedia