Différence entre le séquençage de Sanger et le pyroséquençage
Différence clé - Sanger Sequencing vs pyro-dencing
Le séquençage de l'ADN est très important pour l'analyse de l'ADN, car la connaissance de la disposition des nucléotides correctes sur une région d'ADN particulière révèle de nombreuses informations importantes à ce sujet. Il existe différentes méthodes de séquençage d'ADN. Le séquençage Sanger et le pyroséquençage sont deux méthodes différentes de séquençage d'ADN largement utilisées en biologie moléculaire. La principale différence entre le séquençage de Sanger et le pyroséquençage est que Le séquençage de Sanger utilise des didésoxynucléotides pour terminer la synthèse de l'ADN pour lire la séquence nucléotidique tandis que le pyroséquençage détecte la libération de pyrophosphate en incorporant les nucléotides et en synthétisant la séquence complémentaire pour lire l'ordre précis de la séquence.
CONTENU
1. Aperçu et différence clé
2. Qu'est-ce que le séquençage Sanger
3. Qu'est-ce que le pyrosequençage
4. Comparaison côte à côte - séquençage de Sanger vs pyrosequençage
5. Résumé
Qu'est-ce que le séquençage Sanger?
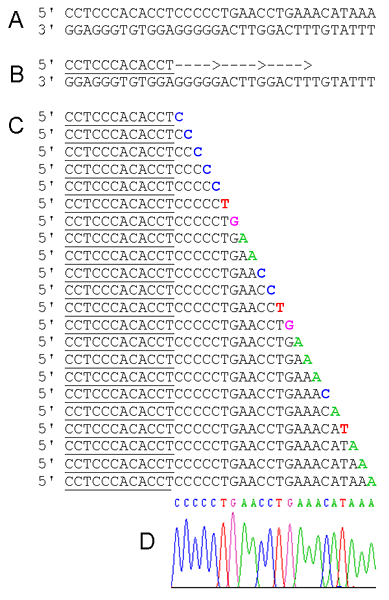
Le séquençage de Sanger est une méthode de séquençage d'ADN de première génération développée par Frederick Sanger et ses collèges en 1977. Il est également connu sous le nom Séquençage de terminaison de la chaîne ou Séquençage de la didésoxy Puisqu'il est basé sur la terminaison de la chaîne par les didésoxynucléotides (DDNTP). Cette méthode a été largement utilisée pendant plus de 30 ans jusqu'à ce que le séquençage de nouvelle génération (NGS) soit développé. La technique de séquençage Sanger a permis la découverte de l'ordre des nucléotides corrects ou la fixation d'un fragment d'ADN particulier. Il est basé sur l'incorporation sélective des DDNTP et la terminaison de la synthèse d'ADN pendant le in vitro Réplication de l'ADN. L'absence de groupes 3 'OH pour poursuivre la formation de liaisons phosphodiester entre les nucléotides adjacentes est une caractéristique unique des DDNTP. Par conséquent, une fois le DDNTP attaché, l'allongement de la chaîne cesse et se termine à partir de ce point. Il existe quatre DDNTPS - DDATP, DDCTP, DDGTP et DDTTP - utilisés dans le séquençage de Sanger. Ces nucléotides arrêtent le processus de réplication de l'ADN lorsqu'ils sont incorporés dans le brin de croissance de l'ADN et entraînent des longueurs variables de l'ADN court. L'électrophorèse de gel capillaire est utilisée pour organiser ces brins d'ADN courts par leurs tailles sur un gel comme le montre la figure 01.
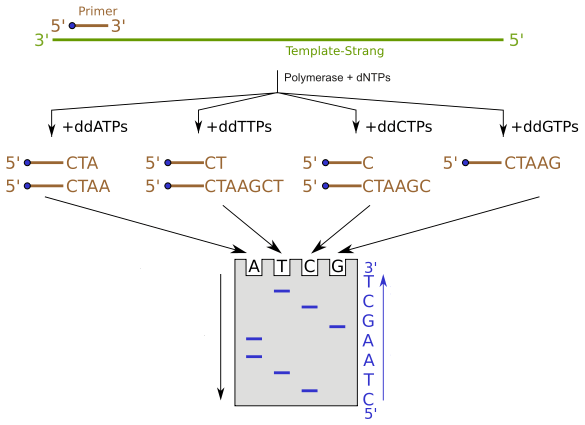
Figure 1: électrophorèse de gel capillaire de l'ADN court synthétisé
Pour in vitro réplication de l'ADN, peu d'exigences devraient être fournies. Ce sont une enzyme de l'ADN polymérase, des modèles d'ADN, des amorces d'oligonucléotides et des désoxynucléotides (DNTP). Dans le séquençage Sanger, la réplication de l'ADN est effectuée dans quatre tubes à essai séparés ainsi que quatre types de DDNTP. Les désoxynucléotides ne sont pas totalement remplacés par les DDNTP respectifs. Un mélange du DNTP particulier (par exemple; DATP + DDATP) est inclus dans le tube et reproduit. Quatre produits de tube séparés sont exécutés sur un gel dans quatre puits séparés. Ensuite, en lisant le gel, la séquence peut être construite comme indiqué sur la figure 02.

Figure 02: séquençage Sanger
Le séquençage Sanger est une technique importante qui aide dans de nombreux domaines de la biologie moléculaire. Le projet du génome humain a été achevé avec succès à l'aide de méthodes basées sur le séquençage Sanger. Le séquençage Sanger est également utile dans le séquençage de l'ADN cible, la recherche sur le cancer et les maladies génétiques, l'analyse de l'expression des gènes, l'identification humaine, la détection des agents pathogènes, le séquençage microbien, etc.
Il existe plusieurs inconvénients du séquençage de Sanger:
- La longueur de l'ADN étant séquencé ne peut pas dépasser 1000 paires de bases
- Un seul brin peut être séquencé à la fois.
- Le processus prend du temps et coûteux.
Par conséquent, de nouvelles techniques avancées de séquençage ont été développées avec le temps pour surmonter ces problèmes. Cependant, le séquençage Sanger est toujours utilisé en raison de ses résultats très précis jusqu'à environ 850 fragments de longueur de paire de bases.
Qu'est-ce que le pyrosequençage?
Le pyroséquençage est une nouvelle technique de séquençage d'ADN basée sur le «séquençage par synthèse». Cette technique repose sur la détection de la libération de pyrophosphate sur l'incorporation de nucléotides. Le processus est utilisé par quatre enzymes différentes: ADN polymère, ATP sulfurylase, luciférase et apyrase et deux substrats adénosine 5 'phosphosulfate (APS) et luciférine.
Le processus commence avec la liaison de l'amorce avec la matrice d'ADN simple brin et l'ADN polymérase commence l'incorporation de nucléotides complémentaires. Lorsque les nucléotides se réunissent (polymérisation de l'acide nucléique), il libère des groupes et de l'énergie pyrophosphate (deux groupes de phosphate liés ensemble). Chaque ajout nucléotidique libère la quantité équimolaire de pyrophosphate. Le pyrophosphate se convertit en ATP par l'ATP sulfurylase en présence de substrat APS. L'ATP généré entraîne la conversion médiée par la luciférase de la luciférine en oxyluciférine, produisant une lumière visible en quantités proportionnelles à la quantité d'ATPS. La lumière est détectée par un dispositif de détection de photons ou par photomultiplier et crée un pyrogramme. L'apyrase dégrade l'ATP et les DNTP non incorporés dans le mélange réactionnel. L'ajout de DNTP est fait une fois à la fois. Étant donné que l'ajout de nucléotide est connu selon l'incorporation et la détection de la lumière, la séquence du modèle peut être déterminée. Le pyrogramme est utilisé pour générer la séquence nucléotidique de l'ADN de l'échantillon comme le montre la figure 03.
Le pyroséquençage est très important dans l'analyse du polymorphisme mononuéotide et le séquençage de courtes étirements d'ADN. La précision élevée, la flexibilité, la facilité d'automatisation et le traitement parallèle sont les avantages du pyroséquençage sur les techniques de séquençage Sanger.
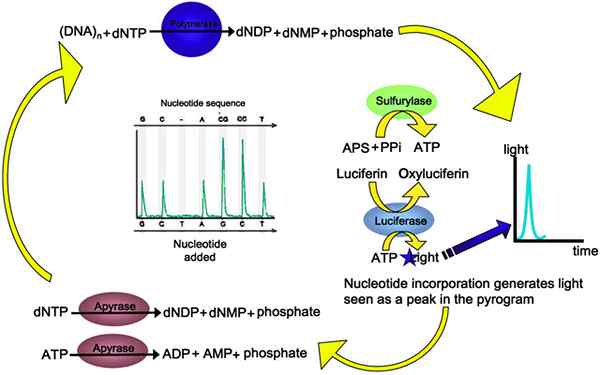
Figure 03: Pyrosequencing
Quelle est la différence entre le séquençage de Sanger et le pyroséquençage?
Séquençage de Sanger vs pyrosequençage | |
| Le séquençage Sanger est une méthode de séquençage d'ADN basée sur l'incorporation sélective des DDNTP par l'ADN polymérase et la terminaison de la chaîne. | Le pyroséquençage est une méthode de séquençage d'ADN basée sur la détection de la libération de pyrophosphate lors de l'incorporation de nucléotides. |
| Utilisation de DDNTP | |
| Les DDNTP sont utilisés pour terminer la réplication de l'ADN | Les DDNTP ne sont pas utilisés. |
| Enzymes impliquées | |
| L'ADN polymérase sont utilisées. | Quatre enzymes sont utilisées: ADN polymérase, ATP sulfurylase, luciférase et apyrase. |
| Substrats utilisés | |
| APS et Luciférine ne sont pas utilisées. | L'adénosine 5 'phosphosulfate (APS) et la luciférine sont utilisées. |
| Température maximale | |
| C'est un processus lent. | C'est un processus rapide. |
Résumé - Sanger Sequencing vs pyrosequencing
Le séquençage Sanger et le pyroséquençage sont deux méthodes de séquençage d'ADN utilisées en biologie moléculaire. Le séquençage de Sanger construit l'ordre des nucléotides en séquence en terminant l'allongement de la chaîne tandis que le pyroséquençage construit l'ordre précis des nucléotides en séquence par incorporation de nucléotides et détectant la libération de pyrophosphates. Par conséquent, la principale différence entre le séquençage de Sanger et le pyroséquençage est que le séquençage de Sanger fonctionne sur le séquençage par terminaison de la chaîne tandis que le pyrosequençage fonctionne sur le séquençage par synthèse.
Référence:
1. Fakruddin, MD et Abhijit Chowdhury. «Pyrosequencing-une alternative au séquençage Sanger traditionnel.»American Journal of Biochemistry and Biotechnology. Publications scientifiques, 02 mars. 2012. la toile. 28 février. 2017.
2. «Sanger séquençage.”Sanger Sequencing - ScienceDirect Sujets. N.p., n.d. la toile. 28 février. 2017
Image gracieuseté:
1. «Didesoxy-méthode» de Christoph Goemans (Modifiziert) - DR. Norman Mauder, auf base einer datei von christoph goemans (cc by-sa 3.0) via Commons Wikimedia
2. «Sanger-dna-seq» par Enzo à la langue polonaise Wikipedia (CC BY-SA 3.0) via Commons Wikimedia
3. «Pyrosequencing» par MicrobiologyBytes (CC BY-SA 2.0) via Flickr